人工智能双参数磁共振成像筛查前列腺癌的诊断准确性:系统综述
- 作者: Kryuchkova O.V.1, Schepkina E.V.2,3,4, Rubtsova N.A.5, Alekseev B.Y.5, Kuznetsov A.I.6, Epifanova S.V.1,3, Zarya E.V.1, Talyshinskii A.E.7
-
隶属关系:
- Central Clinical Hospital, Office of the President of the Russian Federation
- Russian Presidential Academy of National Economy and Public Administration
- Research and Practical Clinical Center for Diagnostics and Telemedical Technologies
- Editorial of the Journal “Pediatria” named after G.N. Speransky
- P.A. Herzen Moscow Oncology Research Institute, Branch National Medical Research Radiological Center
- Moscow Aviation Institute
- Saint Petersburg State University
- 期: 卷 5, 编号 3 (2024)
- 页面: 534-550
- 栏目: 系统评价
- URL: https://journal-vniispk.ru/DD/article/view/310036
- DOI: https://doi.org/10.17816/DD626643
- ID: 310036
如何引用文章
全文:
详细
论证。根据2021年俄罗斯最新公布的数据,将新增40137例前列腺癌病例,在男性人群中仅次于肺癌[2]。
因此,前列腺癌是男性最常见的恶性肿瘤之一。 在这种情况下,准确及时地发现前列腺癌就显得尤为重要。
本系统综述的目的 — 评估在初次就医时确诊前列腺癌构建的预测模型质量。
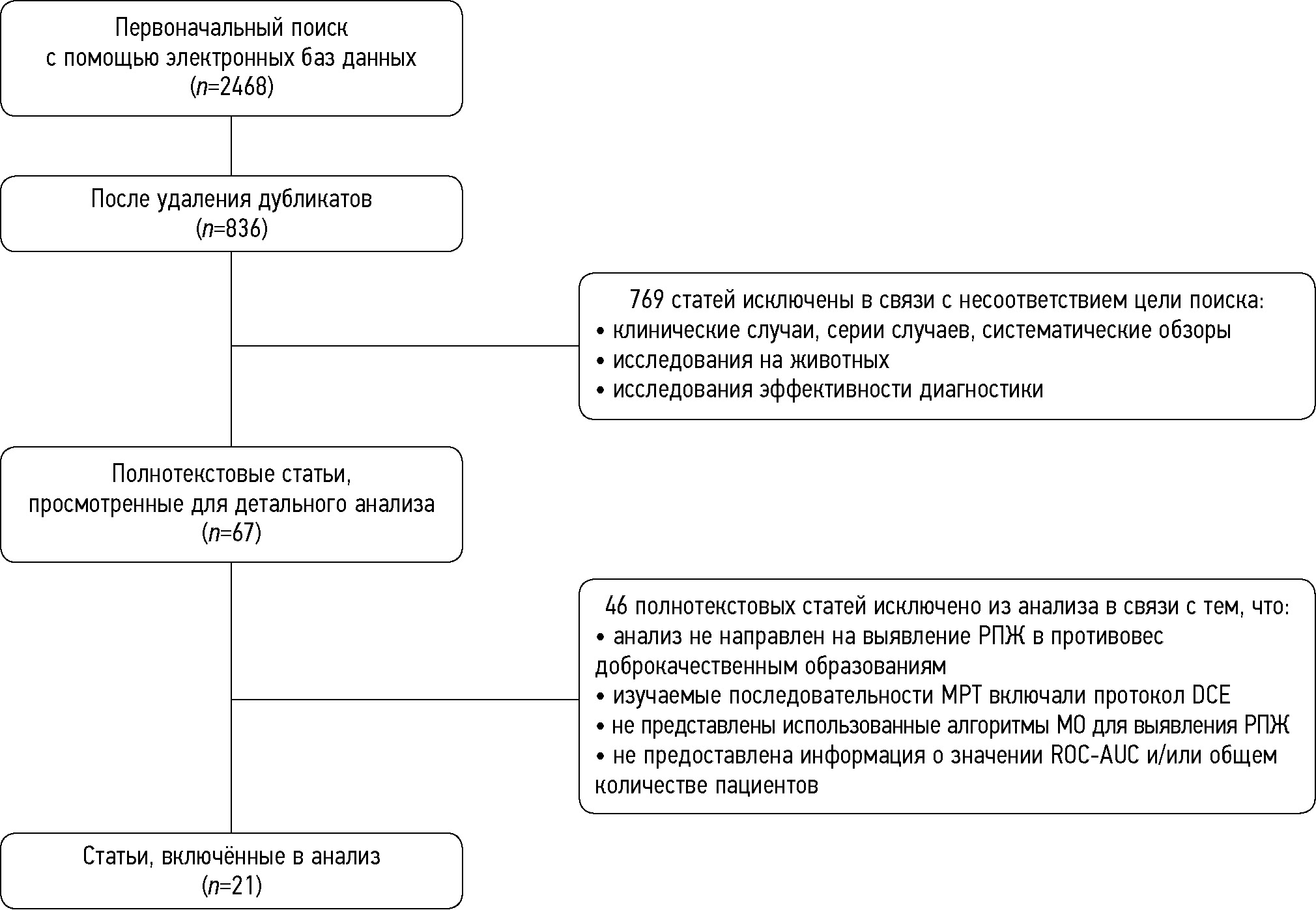
材料和方法。根据PRISMA协议,于2019年1月至2023年9月期间采用既定方法对eLibrary、PubMed、Google Scholar、Web of Science和Research Gate电子数据库中的文献进行了系统检索。 两位作者独立评估了研究对象的纳入与排除。
结果。这项荟萃分析包括21项研究。 共有3630名患者参与,其中 47%患有前列腺癌,53%为良性前列腺增生患者。 患者的平均年龄为67.1岁(年龄范围在36至90岁之间)。81%的研究是基于加权T2成像(T2-WI),57%基于扩散加权成像 (DWI),76%基于表观扩散系数(ADC)。43%的研究为前列腺过渡区(TZ)的增生,33%为前列腺外周区(PZ)。 52%的作者对整个器官进行了研究,而没有划分区域。分析表明,研究人员最常使用以下机器学习 (ML) 算法:MLR(Multiple Logistic Regression)(76%),SVM (Support Vector Machine)(38%) 和 RF(Random Forest) (24%).根据我们研究的文献中描述的73个预测模型的ROC-AUC评估的荟萃分析数据,使用随机效应法,最终ROC-AUC值为0.793[95%CI 0.768;0.818],I2=86.71%,p<0.001。基于T2-WI+ADC序列的模型:(0.860 [95%CI 0.813; 0.907]);以及与《黑盒》原则模型(0.733 [95%CI 0.695; 0.771])相比,最准确的是《白盒》原则模型(0.834 [95%CI0.806;0.861])。用在放射学和临床特征的模型比仅基于放射学特征的模型准确性略高(0.869 [95%CI 0.844; 0.895]vs 0.779 [95%CI 0.751; 0.807])。研究区域(PZ 和/或 TZ)模型的准确性实际上没有区别。
结论。研究结果前景广阔,但临床应用性仍需要医疗机构的专家进行更严格的验证,并在前瞻性研究中进行疗效评估。
作者简介
Oksana V. Kryuchkova
Central Clinical Hospital, Office of the President of the Russian Federation
Email: ovk16@bk.ru
ORCID iD: 0000-0001-6483-2074
SPIN 代码: 2445-3370
MD Cand. Sci. (Medicine)
俄罗斯联邦, MoscowElena V. Schepkina
Russian Presidential Academy of National Economy and Public Administration; Research and Practical Clinical Center for Diagnostics and Telemedical Technologies; Editorial of the Journal “Pediatria” named after G.N. Speransky
编辑信件的主要联系方式.
Email: elenaschepkina@gmail.com
ORCID iD: 0000-0002-2079-1482
SPIN 代码: 2347-9436
Scopus 作者 ID: 57211515165
Researcher ID: IAR-4060-2023
Cand. Sci. (Sociology)
俄罗斯联邦, Moscow; Moscow; MoscowNatalia A. Rubtsova
P.A. Herzen Moscow Oncology Research Institute, Branch National Medical Research Radiological Center
Email: rna17@ya.ru
ORCID iD: 0000-0001-8378-4338
SPIN 代码: 9712-9091
MD, Dr. Sci. (Medicine)
俄罗斯联邦, MoscowBoris Y. Alekseev
P.A. Herzen Moscow Oncology Research Institute, Branch National Medical Research Radiological Center
Email: byalekseev@mail.ru
ORCID iD: 0000-0002-3398-4128
SPIN 代码: 4692-5705
MD, Dr. Sci. (Medicine)
俄罗斯联邦, MoscowAnton I. Kuznetsov
Moscow Aviation Institute
Email: drednout5786@yandex.ru
ORCID iD: 0000-0003-2182-5792
SPIN 代码: 8824-9080
俄罗斯联邦, Moscow
Svetlana V. Epifanova
Central Clinical Hospital, Office of the President of the Russian Federation; Research and Practical Clinical Center for Diagnostics and Telemedical Technologies
Email: svepifanova@yandex.ru
ORCID iD: 0000-0002-7591-5120
SPIN 代码: 9067-5033
MD, Cand. Sci. (Medicine)
俄罗斯联邦, Moscow; MoscowElena V. Zarya
Central Clinical Hospital, Office of the President of the Russian Federation
Email: zaryya@yandex.ru
ORCID iD: 0009-0001-4444-8881
SPIN 代码: 9800-8219
俄罗斯联邦, Moscow
Ali E. Talyshinskii
Saint Petersburg State University
Email: ali-ma@mail.ru
ORCID iD: 0000-0002-3521-8937
SPIN 代码: 7747-0117
MD, Dr. Sci. (Medicine)
俄罗斯联邦, Saint Petersburg参考
- Mottet N, van den Bergh RCN, Briers E, et al. EAU-EANM-ESTRO-ESUR-SIOG Guidelines on Prostate Cancer–2020 Update. Part 1: Screening, Diagnosis, and Local Treatment with Curative Intent. European urology. 2021;79(2):243–262. doi: 10.1016/j.eururo.2020.09.042
- Zdravookhranenie v Rossii, 2021: statisticheskii sbornik. Moscow: Rosstat; 2021.
- Verma S, Rajesh A. A Clinically Relevant Approach to Imaging Prostate Cancer: review. American Journal of Roentgenology. 2011;196(3 Suppl):S1–10 Quiz S11–4. doi: 10.2214/AJR.09.7196
- Girometti R, Giannarini G, Panebianco V, et al. Comparison of different thresholds of PSA density for risk stratification of PI-RADSv2.1 categories on prostate MRI. The British Journal of Radiology. 2022;95(1131):20210886. doi: 10.1259/bjr.20210886
- Niaf E, Lartizien C, Bratan F, et al. Prostate Focal Peripheral Zone Lesions: Characterization at Multiparametric MR Imaging–Influence of a Computer-aided Diagnosis System. Radiology. 2014;271(3):761–769. doi: 10.1148/radiol.14130448
- Drost FJH, Osses DF, Nieboer D, et al. Prostate MRI, with or without MRI-targeted biopsy, and systematic biopsy for detecting prostate cancer. Cochrane Database of Systematic Reviews. 2019;4(4):CD012663. doi: 10.1002/14651858.CD012663.pub2
- Goldenberg SL, Nir G, Salcudean SE. A new era: artificial intelligence and machine learning in prostate cancer. Nature Reviews Urology. 2019;16(7):391–403. doi: 10.1038/s41585-019-0193-3
- Cuocolo R, Cipullo MB, Stanzione A, et al. Machine learning applications in prostate cancer magnetic resonance imaging. European Radiology Experimental. 2019;3(1):35. doi: 10.1186/s41747-019-0109-2
- Ghezzo S, Bezzi C, Presotto L, et al. State of the art of radiomic analysis in the clinical management of prostate cancer: A systematic review. Critical Reviews in Oncology/Hematology. 2022;169:103544. doi: 10.1016/j.critrevonc.2021.103544
- Gelezhe PB, Blokhin IA, Semenov SS, Caruso D. Magnetic resonance imaging radiomics in prostate cancer radiology: what is currently known? Digital Diagnostics. 2021;2(4):441–452. doi: 10.17816/DD70170
- Ferro M, de Cobelli O, Vartolomei MD, et al. Prostate Cancer Radiogenomics–From Imaging to Molecular Characterization. International Journal of Molecular Sciences. 2021;22(18):9971. doi: 10.3390/ijms22189971
- Steyerberg EW, Vickers AJ, Cook NR, et al. Assessing the Performance of Prediction Models. Epidemiology. 2010;21(1):128–138. doi: 10.1097/EDE.0b013e3181c30fb2
- Higgins JPT, Green S, editors. The Cochrane Collaboration. Cochrane Handbook for Systematic Reviews of Interventions [Internet]. England: John Wiley & Sons Ltd. [cited 19 Mar 2020]. Available from: https://training.cochrane.org/handbook
- Higgins JPT, Thompson SG, Deeks JJ, Altman DG. Measuring inconsistency in meta-analyses. BMJ. 2003;327(7414):557–560. doi: 10.1136/bmj.327.7414.557
- DerSimonian R, Laird N. Meta-analysis in clinical trials. Controlled Clinical Trials. 1986;7(3):177–188. doi: 10.1016/0197-2456(86)90046-2
- Woźnicki P, Westhoff N, Huber T, et al. Multiparametric MRI for Prostate Cancer Characterization: Combined Use of Radiomics Model with PI-RADS and Clinical Parameters. Cancers (Basel). 2020;12(7):1767. doi: 10.3390/cancers12071767
- Li M, Yang L, Yue Y, et al. Use of Radiomics to Improve Diagnostic Performance of PI-RADS v2.1 in Prostate Cancer. Frontiers in Oncology. 2021;10:631831. doi: 10.3389/fonc.2020.631831
- Gui S, Lan M, Wang C, et al. Application Value of Radiomic Nomogram in the Differential Diagnosis of Prostate Cancer and Hyperplasia. Frontiers in Oncology. 2022;12:859625. doi: 10.3389/fonc.2022.859625
- Lu Y, Li B, Huang H, et al. Biparametric MRI-based radiomics classifiers for the detection of prostate cancer in patients with PSA serum levels of 4~10 ng/mL. Frontiers in Oncology. 2022;12:1020317. doi: 10.3389/fonc.2022.1020317
- Zhou B, Liu X, Gan H, et al. Differentiation of Prostate Cancer and Stromal Hyperplasia in the Transition Zone With Monoexponential, Stretched-Exponential Diffusion-Weighted Imaging and Diffusion Kurtosis Imaging in a Reduced Number of b Values: Correlation With Whole-Mount Pathology. Journal of Computer Assisted Tomography. 2022;46(4):545–550. doi: 10.1097/RCT.0000000000001314
- Wu M, Krishna S, Thornhill RE, et al. Transition zone prostate cancer: Logistic regression and machine-learning models of quantitative ADC, shape and texture features are highly accurate for diagnosis. Journal of Magnetic Resonance Imaging. 2019;50(3):940–950. doi: 10.1002/jmri.26674
- Zhong JG, Shi L, Liu J, et al. Predicting prostate cancer in men with PSA levels of 4–10 ng/mL: MRI-based radiomics can help junior radiologists improve the diagnostic performance // Scientific reports. 2023;13(1):4846. doi: 10.1038/s41598-023-31869-1
- Ou YC, Chang KH, Tung MC, et al. Building a Nomogram for Prediction of Prostate Cancer in Patients With Preoperatively Suspected Prostate Cancer. Anticancer Research. 2020;40(5):2995–3002. doi: 10.21873/anticanres.14280
- McGarry SD, Bukowy JD, Iczkowski KA, et al. Gleason Probability Maps: A Radiomics Tool for Mapping Prostate Cancer Likelihood in MRI Space. Tomography. 2019;5(1):127–134. doi: 10.18383/j.tom.2018.00033
- Hu L, Zhou DW, Fu CX, et al. Advanced zoomed diffusion-weighted imaging vs. full-field-of-view diffusion-weighted imaging in prostate cancer detection: a radiomic features study. European radiology. 2021;31(3):1760–1769. doi: 10.1007/s00330-020-07227-4
- Ji X, Zhang J, Shi W, et al. Bi-parametric magnetic resonance imaging based radiomics for the identification of benign and malignant prostate lesions: cross-vendor validation. Physical and Engineering Sciences in Medicine. 2021;44(3):745–754. doi: 10.1007/s13246-021-01022-1
- Jin P, Shen J, Yang L, et al. Machine learning-based radiomics model to predict benign and malignant PI-RADS v2.1 category 3 lesions: a retrospective multi-center study. BMC Medical Imaging. 2023;23(1):47. doi: 10.1186/s12880-023-01002-9
- Li S, Zheng T, Fan Z, et al. A dynamic-static combination model based on radiomics features for prostate cancer using multiparametric MRI. Physics in Medicine & Biology. 2023;68(1):015008. doi: 10.1088/1361-6560/aca954
- Ayyad SM, Badawy MA, Shehata M, et al. A New Framework for Precise Identification of Prostatic Adenocarcinoma. Sensors. 2022;22(5):1848. doi: 10.3390/s22051848
- Han L, He G, Mei Y, et al. Combining Magnetic Resonance Diffusion-Weighted Imaging with Prostate-Specific Antigen to Differentiate Between Malignant and Benign Prostate Lesions. Medical Science Monitor. 2022;28:e935307. doi: 10.12659/MSM.935307
- Chen T, Li M, Gu Y, et al. Prostate Cancer Differentiation and Aggressiveness: Assessment With a Radiomic-Based Model vs. PI-RADS v2. Journal of Magnetic Resonance Imaging. 2019;49(3):875–884. doi: 10.1002/jmri.26243
- He D, Wang X, Fu C, et al. MRI-based radiomics models to assess prostate cancer, extracapsular extension and positive surgical margins. Cancer Imaging. 2021;21(1):46. doi: 10.1186/s40644-021-00414-6
- Jamshidi G, Abbasian Ardakani A, Ghafoori M, et al. Radiomics-based machine-learning method to diagnose prostate cancer using mp-MRI: a comparison between conventional and fused models. Magnetic Resonance Materials in Physics, Biology and Medicine. 2022;36(1):55–64. doi: 10.1007/s10334-022-01037-z
- Aussavavirojekul P, Hoonlor A, Srinualnad S. Optimization of clinical risk-factor interpretation and radiological findings with machine learning for PIRADS category 3 patients. Prostate. 2022;82(2):235–244. doi: 10.1002/pros.24266
- Giambelluca D, Cannella R, Vernuccio F, et al. PI-RADS 3 Lesions: Role of Prostate MRI Texture Analysis in the Identification of Prostate Cancer. Current Problems in Diagnostic Radiology. 2021;50(2):175–185. doi: 10.1067/j.cpradiol.2019.10.009
- Viswanath SE, Chirra PV, Yim MC, et al. Comparing radiomic classifiers and classifier ensembles for detection of peripheral zone prostate tumors on T2-weighted MRI: a multi-site study. BMC Medical Imaging. 2019;19(1):22. doi: 10.1186/s12880-019-0308-6
- Chawla NV, Bowyer KW, Hall LO, Kegelmeyer WP. SMOTE: Synthetic Minority Over-sampling Technique. Journal of Artificial Intelligence Research. 2002;16(1):321–357.
- Dai JC, Morgan TN, Goueli R, et al. MRI Features Associated with Histology of Benign Prostatic Hyperplasia Nodules: Generation of a Predictive Model. Journal of Endourology. 2022;36(3):381–386. doi: 10.1089/end.2021.0397
- Liu J, Dong B, Qu W, et al. Using clinical parameters to predict prostate cancer and reduce the unnecessary biopsy among patients with PSA in the gray zone. Scientific reports. 2020;10(1):5157. doi: 10.1038/s41598-020-62015-w
- Zhang L, Tang M, Chen S, et al. A meta-analysis of use of Prostate Imaging Reporting and Data System Version 2 (PI-RADS V2) with multiparametric MR imaging for the detection of prostate cancer. European radiology. 2017;27(12):5204–5214. doi: 10.1007/s00330-017-4843-7
- Zhen L, Liu X, Yegang C, et al. Accuracy of multiparametric magnetic resonance imaging for diagnosing prostate Cancer: a systematic review and meta-analysis. BMC Cancer. 2019;19(1):1244. doi: 10.1186/s12885-019-6434-2
补充文件