Monitoring the spread of the SARS-CoV-2 (Coronaviridae: Coronavirinae: Betacoronavirus; Sarbecovirus) variants in the Moscow region using targeted high-throughput sequencing
- Authors: Borisova N.I.1, Kotov I.A.1,2, Kolesnikov A.A.1, Kaptelova V.V.1, Speranskaya A.S.1, Kondrasheva L.Y.1, Tivanova E.V.1, Khafizov K.F.1, Akimkin V.G.1
-
Affiliations:
- FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- FSAEI HE «Moscow Institute of Physics and Technology (National Research University)»
- Issue: Vol 66, No 4 (2021)
- Pages: 269-278
- Section: ORIGINAL RESEARCHES
- URL: https://journal-vniispk.ru/0507-4088/article/view/118179
- DOI: https://doi.org/10.36233/0507-4088-72
- ID: 118179
Cite item
Abstract
Introduction. Since the outbreak of the COVID-19 pandemic caused by SARS-CoV-2 novel coronavirus, the international community has been concerned about the emergence of mutations altering some biological properties of the pathogen like increasing its infectivity or virulence. Particularly, since the end of 2020, several variants of concern have been identified around the world, including Alpha (B.1.1.7), Beta (B.1.351), Gamma (P.1), and Delta (B.1.617.2). However, the existing mechanism of detecting important mutations are not always effective enough, since only a relatively small part of all pathogen samples can be examined by whole genome sequencing due to its high cost.
Material and methods. In this study, we have designed special primer panel and used it for targeted highthroughput sequencing of several significant S-gene (spike) regions of SARS-CoV-2. The Illumina platform averaged approximately 50,000 paired-end reads with a length of ≥150 bp per sample. This method was used to examine 579 random samples obtained from COVID-19 patients in Moscow and the Moscow region from February to June 2021.
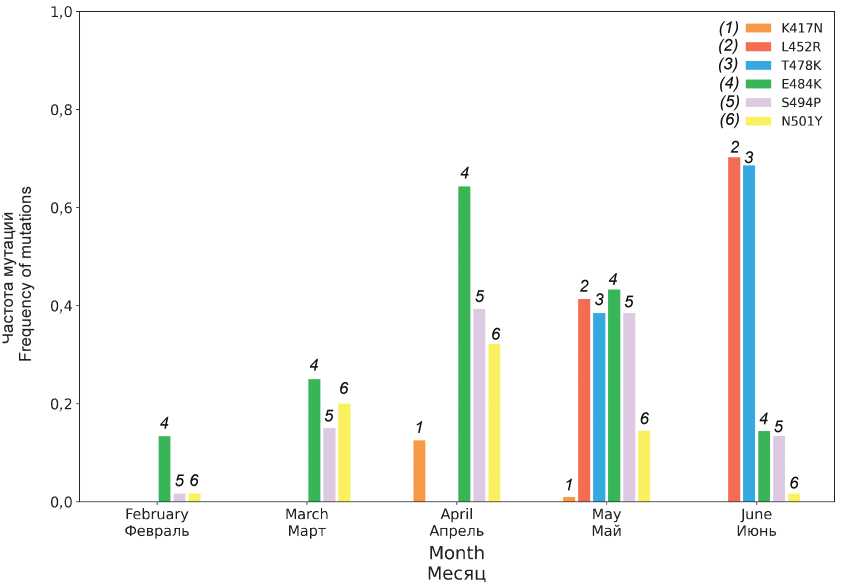
Results. This study demonstrated the dynamics of distribution of several SARS-CoV-2 strains and its some single mutations. It was found that the Delta strain appeared in the region in May 2021, and became prevalent in June, partially displacing other strains.
Discussion. The obtained results provide an opportunity to assign the viral samples to one of the strains, including the previously mentioned in time- and cost-effective manner. The approach can be used for standardization of the procedure of searching for mutations in individual regions of the SARS-CoV-2 genome. It allows to get a more detailed data about the epidemiological situation in a region.
Full Text
##article.viewOnOriginalSite##About the authors
N. I. Borisova
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Author for correspondence.
ORCID iD: 0000-0002-9672-0648
Moscow, 111123, Russia
Russian FederationI. A. Kotov
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor); FSAEI HE «Moscow Institute of Physics and Technology (National Research University)»
ORCID iD: 0000-0003-2416-5689
Moscow, 111123, Russia
Moscow, 111123, Russia
Russian FederationA. A. Kolesnikov
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0003-3480-953X
Moscow, 111123, Russia
Russian FederationV. V. Kaptelova
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0003-0952-0830
Moscow, 111123, Russia
Russian FederationA. S. Speranskaya
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0001-6326-1249
Moscow, 111123, Russia
Russian FederationL. Yu. Kondrasheva
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-0147-4262
Moscow, 111123, Russia
Russian FederationE. V. Tivanova
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0003-1286-2612
Moscow, 111123, Russia
Russian FederationK. F. Khafizov
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: kkhafizov@gmail.com
ORCID iD: 0000-0001-5524-0296
Kamil F. Khafizov, Ph.D. (Biol.), Head of the Scientific Group for the Development of New Diagnostic Methods, Department of Molecular Diagnostics and Epidemiology
Moscow, 111123, Russia
Russian FederationV. G. Akimkin
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0003-4228-9044
Moscow, 111123, Russia
Russian FederationReferences
- Zhou P., Yang X.L., Wang X.G., Hu B., Zhang L., Zhang W., et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020; 579(7798): 270–3. https://doi.org/10.1038/s41586-020-2012-7
- COVID-19 data in motion. Available at: https://coronavirus.jhu.edu (accessed July 24, 2021).
- Chen P., Nirula A., Heller B., Gottlieb R.L., Boscia J., Morris J., et al. SARS-CoV-2 neutralizing antibody LY-CoV555 in outpatients with Covid-19. N. Engl. J. Med. 2021; 384(3): 229–37. https://doi.org/10.1056/nejmoa2029849
- Weinreich D.M., Sivapalasingam S., Norton T., Ali S., Gao H., Bhore R., et al. REGN-COV2, a neutralizing antibody cocktail, in outpatients with Covid-19. N. Engl. J. Med. 2021; 384(3): 238–51. https://doi.org/10.1056/nejmoa2035002
- Baden L.R., El Sahly H.M., Essink B., Kotloff K., Frey S., Novak R., et al. Efficacy and safety of the mRNA-1273 SARS-CoV-2 vaccine. N. Engl. J. Med. 2021; 384(5): 403–16. https://doi.org/10.1056/nejmoa2035389
- Polack F.P., Thomas S.J., Kitchin N., Absalon J., Gurtman A., Lockhart S., et al. Safety and efficacy of the BNT162b2 mRNA Covid-19 Vaccine. N. Engl. J. Med. 2020; 383(27): 2603–15. https://doi.org/10.1056/nejmoa2034577
- Jones I., Roy P. Sputnik V COVID-19 vaccine candidate appears safe and effective. Lancet. 2021; 397(10275): 642–3. https://doi.org/10.1016/s0140-6736(21)00191-4
- Рыжиков А.Б., Рыжиков Е.А., Богрянцева М.П., Усова С.В., Даниленко Е.Д., Нечаева Е.А., и др. Простое слепое плацебо-контролируемое рандомизированное исследование безопасности, реактогенности и иммуногенности вакцины «ЭпиВакКорона» для профилактики COVID-19 на добровольцах в возрасте 18–60 лет (фаза I–II). Инфекция и иммунитет. 2021; 11(2): 283–96. https://doi.org/10.15789/2220-7619-ASB-1699
- About Variants of the Virus that Causes COVID-19. Available at: https://www.cdc.gov/coronavirus/2019-ncov/Transmission/variant.html (accessed July 26, 2021).
- Wang W.B., Liang Y., Jin Y.Q., Zhang J., Su J.G., Li Q.M. E484K mutation in SARS-CoV-2 RBD enhances binding affinity with hACE2 but reduces interactions with neutralizing antibodies and nanobodies: binding free energy calculation studies. bioRxiv. 2021; Preprint. https://doi.org/10.1101/2021.02.17.431566
- Garcia-Beltran W.F., Lam E.C., St. Denis K., Nitido A.D., Garcia Z.H., Hauser B.M., et al. Circulating SARS-CoV-2 variants escape neutralization by vaccine-induced humoral immunity. medRxiv. 2021; Preprint. https://doi.org/10.1101/2021.02.14.21251704
- Liu H., Wei P., Zhang Q., Chen Z., Aviszus K., Downing W., et al. 501Y.V2 and 501Y.V3 variants of SARS-CoV-2 lose binding to bamlanivimab in vitro. MAbs. 2021; 13(1): 1919285. https://doi.org/10.1080/19420862.2021.1919285
- Yuan M., Huang D., Lee C.D., Wu N.C., Jackson A.M., Zhu X., et al. Structural and functional ramifications of antigenic drift in recent SARS-CoV-2 variants. Science. 2021; eabh1139. https://doi.org/10.1126/science.abh1139
- Ikegame S., Siddiquey M.N.A., Hung C.-T., Haas G., Brambilla L., Oguntuyo K.Y., et al. Neutralizing activity of Sputnik V vaccine sera against SARS-CoV-2 variants. medRxiv. 2021.03.31.21254660. doi: https://doi.org/10.1101/2021.03.31.21254660
- Gard N., Buzko O., Spilman P., Niazi K., Rabizadeh S., Soon- Shiong P. Molecular dynamic simulation reveals E484K mutation enhances spike RBD-ACE2 affinity and the combination of E484K, K417N and N501Y mutations (501Y.V2 variant) induces conformational change greater than N501Y mutant alone, potentially resulting in an escape mutant bioRxiv. 2021.01.13.426558. doi: https://doi.org/10.1101/2021.01.13.426558
- Tian F., Tong B., Sun L., Shi S., Zheng B., Wang Z., et al. Mutation N501Y in RBD of spike protein strengthens the interaction between COVID-19 and its receptor ACE2. bioRxiv. 2021; Preprint. https://doi.org/10.1101/2021.02.14.431117
- Хафизов К.Ф., Петров В.В., Красовитов К.В., Золкина М.В., Акимкин В.Г. Экспресс-диагностика новой коронавирусной инфекции с помощью реакции петлевой изотермической амплификации. Вопросы вирусологии. 2021; 66(1): 17–28. https://doi.org/10.36233/0507-4088-42
- Gladkikh A., Dolgova A., Dedkov V., Sbarzaglia V., Kanaeva O., Popova A., et al. Characterization of a novel SARS-CoV-2 genetic variant with distinct spike protein mutations. Viruses. 2021; 13(6): 1029. https://doi.org/10.3390/v13061029
- Klink G.V., Safina K.R., Garushyants S.K., Moldovan M., Nabieva E., Komissarov A.B., et al. Spread of endemic SARSCoV-2 lineages in Russia. medRxiv. 2021; Preprint. https://doi.org/10.1101/2021.05.25.21257695
- Komissarov A.B., Safina K.R., Garushyants S.K., Fadeev A.V., Sergeeva M.V., Ivanova A.A., et al. Genomic epidemiology of the early stages of the SARS-CoV-2 outbreak in Russia. Nat. Commun. 2021; 12(1): 649. https://doi.org/10.1038/s41467-020-20880-z
- Long S.W., Olsen R.J., Christensen P.A., Subedi S., Olson R., Davis J.J., et al. Sequence Analysis of 20,453 Severe Acute Respiratory Syndrome Coronavirus 2 Genomes from the Houston Metropolitan Area Identifies the Emergence and Widespread Distribution of Multiple Isolates of All Major Variants of Concern. Am. J. Pathol. 2021; 191(6): 983–92. https://doi.org/10.1016/j.ajpath.2021.03.004
- Altschul S.F., Gish W., Miller W., Myers E.W., Lipman D.J. Basic local alignment search tool. J. Mol. Biol. 1990; 215(3): 403–10. https://doi.org/10.1016/s0022-2836(05)80360-2
- Li H., Durbin R. Fast and accurate short read alignment with Burrows– Wheeler transform. Bioinformatics. 2009; 25(14): 1754–60. https://doi.org/10.1093/bioinformatics/btp324
- Bushnell B., Rood J., Singer E. BBMerge – Accurate paired shotgun read merging via overlap. PLoS One. 2017; 12(10): e0185056. https://doi.org/10.1371/journal.pone.0185056
- Poplin R., Ruano-Rubio V., DePristo M.A., Fennell T.J., Carneiro M.O., Van der Auwera G.A., et al. Scaling accurate genetic variant discovery to tens of thousands of samples. bioRxiv. 2018; 201178. doi: https://doi.org/10.1101/201178
- Humphrey W., Dalke A., Schulten K. VMD: visual molecular dynamics. J. Mol. Graph. 1996; 14(1): 33–8. https://doi.org/10.1016/0263-7855(96)00018-5
- Lv Z., Deng Y.Q., Ye Q., Cao L., Sun C.Y., Fan C., et al. Structural basis for neutralization of SARS-CoV-2 and SARS-CoV by a potent therapeutic antibody. Science. 2020; 369(6510): 1505–9. https://doi.org/10.1126/science.abc5881
- Davies N.G., Abbott S., Barnard R.C., Jarvis C.I., Kucharski A.J., Munday J.D., et al. Estimated transmissibility and impact of SARSCoV-2 lineage B.1.1.7 in England. Science. 2021; 372(6538): eabg3055. https://doi.org/10.1126/science.abg3055
- Expert comment on the ‘Delta plus’ variant (B.1.617.2 with the addition of K417N mutation). Available at: https://www.sciencemediacentre.org/expert-comment-on-the-delta-plus-variant-b-1-617-2-with-theaddition-of-k417n-mutation/ (accessed July 26, 2021).
- Weekly epidemiological update on COVID-19 – 22 June 2021. Available at: https://www.who.int/publications/m/item/weekly-epidemiological-update-on-covid-19---22-june-2021 (accessed July 24, 2021).
Supplementary files