Analysis of Tat protein characteristics in human immunodeficiency virus type 1 sub-subtype A6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1)
- Authors: Kuznetsova A.I.1, Gromov K.B.1,2, Kireev D.E.2, Shlykova A.V.2, Lopatukhin A.E.2, Kazennova E.V.1, Lebedev A.V.1, Tumanov A.S.1, Kim K.V.1, Bobkova M.R.1
-
Affiliations:
- D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
- FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- Issue: Vol 66, No 6 (2021)
- Pages: 452-464
- Section: ORIGINAL RESEARCHES
- URL: https://journal-vniispk.ru/0507-4088/article/view/118222
- DOI: https://doi.org/10.36233/0507-4088-83
- ID: 118222
Cite item
Abstract
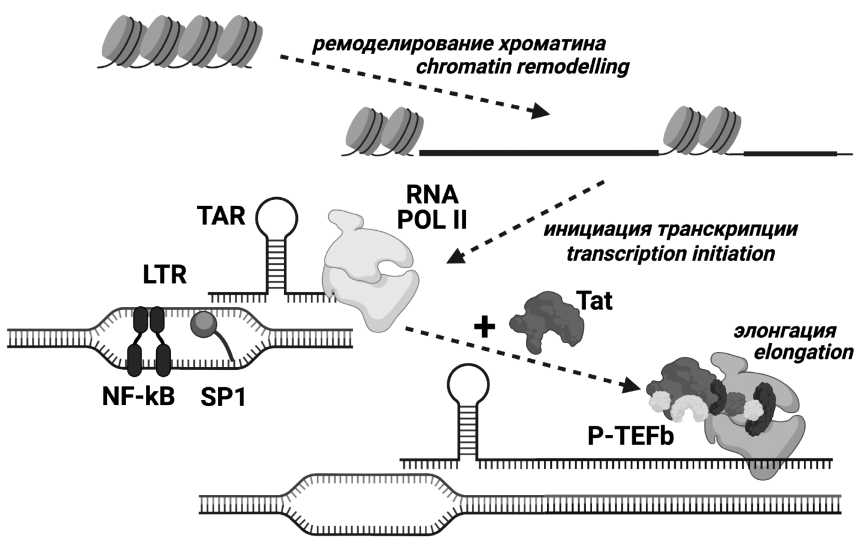
Introduction. Tat protein is a major factor of HIV (human immunodeficiency virus) transcription regulation and has other activities. Tat is characterized by high variability, with some amino acid substitutions, including subtypespecific ones, being able to influence on its functionality. HIV type 1 (HIV-1) sub-subtype A6 is the most widespread in Russia. Previous studies of the polymorphisms in structural regions of the A6 variant have shown numerous characteristic features; however, Tat polymorphism in A6 has not been studied.
Goals and tasks. The main goal of the work was to analyze the characteristics of Tat protein in HIV-1 A6 variant, that is, to identify substitutions characteristic for A6 and A1 variants, as well as to compare the frequency of mutations in functionally significant domains in sub-subtype A6 and subtype B.
Material and methods. The nucleotide sequences of HIV-1 sub-subtypes A6, A1, A2, A3, A4, subtype B and the reference nucleotide sequence were obtained from the Los Alamos international database.
Results and discussion. Q54H and Q60H were identified as characteristic substitutions. Essential differences in natural polymorphisms between sub-subtypes A6 and A1 have been demonstrated. In the CPP-region, there were detected mutations (R53K, Q54H, Q54P, R57G) which were more common in sub-subtype A6 than in subtype B.
Conclusion. Tat protein of sub-subtype A6 have some characteristics that make it possible to reliably distinguish it from other HIV-1 variants. Mutations identified in the CPP region could potentially alter the activity of Tat. The data obtained could form the basis for the drugs and vaccines development.
Full Text
##article.viewOnOriginalSite##About the authors
A. I. Kuznetsova
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
Author for correspondence.
Email: a-myznikova@list.ru
ORCID iD: 0000-0001-5299-3081
Anna I. Kuznetsova, Ph.D. (Biol.), Senior Researcher, Laboratory of T-lymphotropic viruses
123098, Moscow, Russia
Russian FederationK. B. Gromov
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia; FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-9316-1975
123098, Moscow, Russia
Moscow, 111123, Russia
Russian FederationD. E. Kireev
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-7896-2379
Moscow, 111123, Russia
Russian FederationA. V. Shlykova
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-1390-8021
Moscow, 111123, Russia
Russian FederationA. E. Lopatukhin
FSBI «Central Research Institute for Epidemiology» of the Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
ORCID iD: 0000-0002-2826-699X
Moscow, 111123, Russia
Russian FederationE. V. Kazennova
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0002-7912-4270
123098, Moscow, Russia
Russian FederationA. V. Lebedev
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0001-6787-9345
123098, Moscow, Russia
Russian FederationA. S. Tumanov
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0002-6221-5678
123098, Moscow, Russia
Russian FederationK. V. Kim
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0002-4150-2280
123098, Moscow, Russia
Russian FederationM. R. Bobkova
D.I. Ivanovsky Institute of Virology FSBI «National Research Centre for Epidemiology and Microbiology named after the honorary academician N.F. Gamaleya» of the Ministry of Health of Russia
ORCID iD: 0000-0001-5481-8957
123098, Moscow, Russia
Russian FederationReferences
- Campbell G.R., Loret E.P. What does the structure-function relationship of the HIV-1 Tat protein teach us about developing an AIDS vaccine? Retrovirology. 2009; 6: 50. https://doi.org/10.1186/1742-4690-6-50
- Jin H., Li D., Lin M.H., Li L., Harrich D. Tat-based therapies as an adjuvant for an HIV-1 functional cure. Viruses. 2020; 12(4): 415. https://doi.org/10.3390/v12040415
- Asamitsu K., Fujinaga K., Okamoto T. HIV tat/P-TEFb interaction: a potential target for novel anti-HIV therapies. Molecules. 2018; 23(4): 933. https://doi.org/10.3390/molecules23040933
- Лаповок И.А., Лопатухин А.Э., Киреев Д.Е., Казеннова Е.В., Лебедев А.В., Бобкова М.Р., и др. Молекулярно-эпидемиологический анализ вариантов ВИЧ-1, циркулировавших в Рос- сии в 1987–2015 гг. Терапевтический архив. 2017; 89(11): 44–9. https://doi.org/10.17116/terarkh2017891144-49
- Громов К.Б., Киреев Д.Е., Мурзакова А.В., Лопатухин А.Э., Казеннова Е.В., Бобкова М.Р. Анализ полиморфизма белка Nef вариантов ВИЧ-1 (Human immunodeficiency virus-1, Lentivirus, Orthoretrovirinae, Retroviridae), циркулирующих в странах бывшего СССР. Вопросы вирусологии. 2019; 64(6): 281–90. https://doi.org/10.36233/0507-4088-2019-64-6-281-290
- Rosen C.A. Tat and Rev: positive modulators of human immunodeficiency virus gene expression. Gene Expr. 1991; 1(2): 85–90.
- Clark E., Nava B., Caputi M. Tat is a multifunctional viral protein that modulates cellular gene expression and functions. Oncotarget. 2017; 8(16): 27569–81. https://doi.org/10.18632/oncotarget.15174
- Ajasin D., Eugenin E.A. HIV-1 Tat: Role in bystander toxicity. Front. Cell. Infect. Microbiol. 2020; 10: 61. https://doi.org/10.3389/fcimb.2020.00061
- Spector C., Mele A.R., Wigdahl B., Nonnemacher M.R. Genetic variation and function of the HIV-1 Tat protein. Med. Microbiol. Immunol. 2019; 208(2): 131–69. https://doi.org/10.1007/s00430-019-00583-z
- Asamitsu K., Okamoto T. The Tat/P-TEFb protein-protein interaction determining transcriptional activation of HIV. Curr. Pharm. Des. 2017; 23(28): 4091–7. https://doi.org/10.2174/1381612823666170710164148
- Nekhai S., Jeang K.T. Transcriptional and post-transcriptional regulation of HIV-1 gene expression: role of cellular factors for Tat and Rev. Future Microbiol. 2006; 1(4): 417–26. https://doi.org/10.2217/17460913.1.4.417
- Vardabasso C., Manganaro L., Lusic M., Marcello A., Giacca M. The histone chaperone protein nucleosome assembly protein-1 (hNAP-1) binds HIV-1 Tat and promotes viral transcription. Retrovirology. 2008; 5: 8. https://doi.org/10.1186/1742-4690-5-8
- Kamori D., Ueno T. HIV-1 Tat and viral latency: What we can learn from naturally occurring sequence variations. Front. Microbiol. 2017; 8: 80. https://doi.org/10.3389/fmicb.2017.00080
- Rodríguez-Mora S., Mateos E., Moran M., Martín M.Á., López J.A., Calvo E., et al. Intracellular expression of Tat alters mitochondrial functions in T cells: a potential mechanism to understand mitochondrial damage during HIV-1 replication. Retrovirology. 2015; 12: 78. https://doi.org/10.1186/s12977-015-0203-3
- Darbinian N., Darbinyan A., Merabova N., Selzer M.E., Amini S. HIV-1 and HIV-1-Tat induce mitochondrial DNA damage in human neurons. HIV AIDS. 2020; 6(1): 176. https://doi.org/10.16966/2380-5536.176
- Kim J., Kim Y.S. Effect of HIV-1 Tat on the formation of the mitotic spindle by interaction with ribosomal protein S3. Sci. Rep. 2018; 8(1): 8680. https://doi.org/10.1038/s41598-018-27008-w
- Debaisieux S., Rayne F., Yezid H., Beaumelle B. The ins and outs of HIV-1 Tat. Traffic. 2012; 13(3): 355–63. https://doi.org/10.1111/j.1600-0854.2011.01286.x
- Ruiz A.P., Ajasin D.O., Ramasamy S., DesMarais V., Eugenin E.A., Prasad V.R. A naturally occurring polymorphism in the HIV-1 Tat basic domain inhibits uptake by bystander cells and leads to reduced neuroinflammation. Sci. Rep. 2019; 9(1): 3308. https://doi.org/10.1038/s41598-019-39531-5
- Wenzel E.D., Avdoshina V., Mocchetti I. HIV-associated neurodegeneration: exploitation of the neuronal cytoskeleton. Neurovirol. 2019; 25(3): 301–12. https://doi.org/10.1007/s13365-019-00737-y
- Simenauer A., Nozik-Grayck E., Cota-Gomez A. The DNA damage response and HIV-associated pulmonary arterial hypertension. Int. J. Mol. Sci. 2020; 21(9): 3305. https://doi.org/10.3390/ijms21093305
- Anand A.R., Rachel G., Parthasarathy D. HIV proteins and endothelial dysfunction: implications in cardiovascular disease. Front. Cardiovasc. Med. 2018; 5: 185. https://doi.org/10.3389/fcvm.2018.00185
- Brailoiu E., Deliu E., Sporici R.A., Benamar K., Brailoiu G.C. HIV- 1-Tat excites cardiac parasympathetic neurons of nucleus ambiguus and triggers prolonged bradycardia in conscious rats. Am. J. Physiol. Regul. Integr. Comp. Physiol. 2014; 306(11): R814–22. https://doi.org/10.1152/ajpregu.00529.2013
- Jiang Y., Chai L., Fasae M.B., Bai Y. The role of HIV Tat protein in HIV-related cardiovascular diseases. J. Transl. Med. 2018; 16(1):121. https://doi.org/10.1186/s12967-018-1500-0
- Isaguliants M., Bayurova E., Avdoshina D., Kondrashova A., Chiodi F., Palefsky J.M. Oncogenic effects of HIV-1 proteins, mechanisms behind. Cancers. 2021; 13(2): 305. https://doi.org/10.3390/cancers13020305
- Li L., Dahiya S., Kortagere S., Aiamkitsumrit B., Cunningham D., Pirrone V., et al. Impact of Tat genetic variation on HIV-1 disease. Adv. Virol. 2012; 2012: 123605. https://doi.org/10.1155/2012/123605
- López-Huertas M.R., Mateos E., del Cojo M.S., Gómez-Esquer F., Díaz-Gil G., Rodríguez-Mora S., et al. The presence of HIV-1 Tat protein second exon delays Fas protein-mediated apoptosis in CD4+ T Lymphocytes: a potential mechanism for persistent viral production. J. Biol. Chem. 2013; 288(11): 7626–44. https://doi.org/10.1074/jbc.M112.408294
- Mishra M., Vetrivel S., Siddappa N.B., Ranga U., Seth P. Clade-specific differences in neurotoxicity of human immunodeficiency virus-1 B and C Tat of human neurons: significance of dicysteine C30C31 motif. Ann. Neurol. 2008; 63(3): 366–76. https://doi.org/10.1002/ana.21292
- Rao V.R., Sas A.R., Eugenin E.A., Siddapa N.B., Bimonte-Nelson H., Berman J.W., et al. HIV-1 clade-specific differences in the induction of neuropathogenesis. J. Neurosci. 2008; 28(40): 10010–6. https://doi.org/10.1523/JNEUROSCI.2955-08.2008
- Rao V.R., Neogi U., Talboom J.S., Padilla L., Rahman M., Fritz- French C., et al. Clade C HIV-1 isolates circulating in Southern Africa exhibit a greater frequency of dicysteine motif-containing Tat variants than those in Southeast Asia and cause increased neurovirulence. Retrovirology. 2013; 10: 61. https://doi.org/10.1186/1742-4690-10-61
- Paul R.H., Joska J.A., Woods C., Seedat S., Engelbrecht S., Hoare J., et al. Impact of the HIV Tat C30C31S dicysteine substitution on neuropsychological function in patients with clade C disease. J. Neurovirol. 2014; 20(6): 627–35. https://doi.org/10.1007/s13365-014-0293-z
- Vivès E., Brodin P., Lebleu B. A truncated HIV-1 Tat protein basic domain rapidly translocates through the plasma membrane and accumulates in the cell nucleus. J. Biol. Chem. 1997; 272(25): 16010–7. https://doi.org/10.1074/jbc.272.25.16010
- Ziegler A., Seelig J. Interaction of the protein transduction domain of HIV-1 TAT with heparan sulfate: binding mechanism and thermodynamic parameters. Biophys. J. 2004; 86(1 Pt. 1): 254–63. https://doi.org/10.1016/s0006-3495(04)74101-6
- Ziegler A., Nervi P., Dürrenberger M., Seelig J. The cationic cell-penetrating peptide CPPTAT derived from the HIV-1 protein TAT is rapidly transported into living fibroblasts: optical, biophysical, and metabolic evidence. Biochemistry. 2005; 44(1): 138–8. https://doi.org/10.1021/bi0491604
- de Almeida S.M., Rotta I., Vidal L.R.R., Dos Santos J.S., Nath A., Johnson K., et al. HIV-1C and HIV-1B Tat protein polymorphism in Southern Brazil. J. Neurovirol. 2021; 27(1): 126–36. https://doi.org/10.1007/s13365-020-00935-z
- Bobkov A., Kazennova E., Selimova L., Bobkova M., Khanina T., Ladnaya N., et al. A sudden epidemic of HIV type 1 among injecting drug users in the former Soviet Union: identification of subtype A, subtype B, and novel gagA/envB recombinants. AIDS Res. Hum. Retroviruses. 1998; 14(8): 669–76. https://doi.org/10.1089/aid.1998.14.669
- Lebedev A., Lebedeva N., Moskaleychik F., Pronin A., Kazennova E., Bobkova M. Human immunodeficiency virus-1 diversity in the Moscow Region, Russia: Phylodynamics of the most common subtypes. Front. Microbiol. 2019; 10: 320. https://doi.org/10.3389/fmicb.2019.00320
- Казеннова Е.В., Лаповок И.А., Лага В.Ю., Васильев А.В., Бобкова М.Р. Естественные полиморфизмы гена pol варианта ВИЧ-1 IDU-A. ВИЧ-инфекция и иммуносупрессии. 2012; 4(4):44–51.
- Васильев А.В., Казеннова Е.В., Бобкова М.Р. Предсказание фенотипа R5/X4 вариантов ВИЧ-1, циркулирующих в России, с использованием компьютерных методов. Вопросы вирусологии. 2009; 54(3): 17–20.
- Казеннова Е.В., Васильев А.В., Бобкова М.Р. Прогноз эффективности применения препарата Бевиримат для лечения ВИЧ-инфекции в России. Вопросы вирусологии. 2010; 55(3): 37–41.
- Lapovok I., Laga V., Kazennova E., Bobkova M. HIV type 1 integrase natural polymorphisms in viral variants circulating in FSU countries. Curr. HIV Res. 2017; 15(5): 318–26. https://doi.org/10.2174/1570162X15666170815162052
- Shafer R.W., Rhee S.Y., Pillay D., Miller V., Sandstrom P., Schapiro J.M., et al. HIV protease and reverse transcriptase mutations for drug resistance surveillance. AIDS. 2007; 21(2): 215–23. https://doi.org/10.1097/qad.0b013e328011e691
- Jin H., Sun Y., Li D., Lin M.H., Lor M., Rustanti L., et al. Strong in vivo inhibition of HIV-1 replication by Nullbasic, a Tat mutant. mBio. 2019; 10(4): e01769–19. https://doi.org/10.1128/mBio.01769-19
- Leoz M., Kukanja P., Luo Z., Huang F., Cary D.C., Peterlin B.M., et al. HEXIM1-Tat chimera inhibits HIV-1 replication. PLoS Pathog. 2018; 14(11): e1007402. https://doi.org/10.1371/journal.ppat.1007402
- Mediouni S., Chinthalapudi K., Ekka M.K., Usui I., Jablonski J.A., Clementz M.A., et al. Didehydro-cortistatin A inhibits HIV-1 by specifically binding to the unstructured basic region of Tat. mBio. 2019; 10(1): e02662–18. https://doi.org/10.1128/mBio.02662-18
- Wan Z., Chen X. Triptolide inhibits human immunodeficiency virus type 1 replication by promoting proteasomal degradation of Tat protein. Retrovirology. 2014; 11: 88. https://doi.org/10.1186/s12977-014-0088-6
- Sgadari C., Monini P., Tripiciano A., Picconi O., Casabianca A., Orlandi C., et al. Continued decay of HIV proviral DNA upon vaccination with HIV-1 Tat of subjects on long-term ART: An 8-year follow-up study. Front. Immunol. 2019; 10: 233. https://doi.org/10.3389/fimmu.2019.00233
- Loret E.P., Darque A., Jouve E., Loret E.A., Nicolino-Brunet C., Morange S., et al. Intradermal injection of a Tat Oyi-based therapeutic HIV vaccine reduces of 1.5 log copies/mL the HIV RNA rebound median and no HIV DNA rebound following cart interruption in a phase I/II randomized controlled clinical trial. Retrovirology. 2016; 13: 21. https://doi.org/10.1186/s12977-016-0251-3
Supplementary files