Genetic diversity of the human immunodeficiency virus (HIV-1) in the Kaliningrad region
- Authors: Shchemelev A.N.1, Semenov A.V.2, Ostankova Y.V.1, Naidenova E.V.3, Zueva E.B.1, Valutite D.E.1, Churina M.A.4, Virolainen P.A.1, Totolian A.A.1
-
Affiliations:
- FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- Ekaterinburg Research Institute of Viral Infections of the Federal Research Institute, State Research Center for Virology and Biotechnology “Vector” of the Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- FSSI Russian Research Anti-Plague Institute «Microbe» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
- St. Petersburg GBUZ «Botkin Clinical Infectious Diseases Hospital»
- Issue: Vol 67, No 4 (2022)
- Pages: 310-321
- Section: ORIGINAL RESEARCHES
- URL: https://journal-vniispk.ru/0507-4088/article/view/118231
- DOI: https://doi.org/10.36233/0507-4088-119
- ID: 118231
Cite item
Abstract
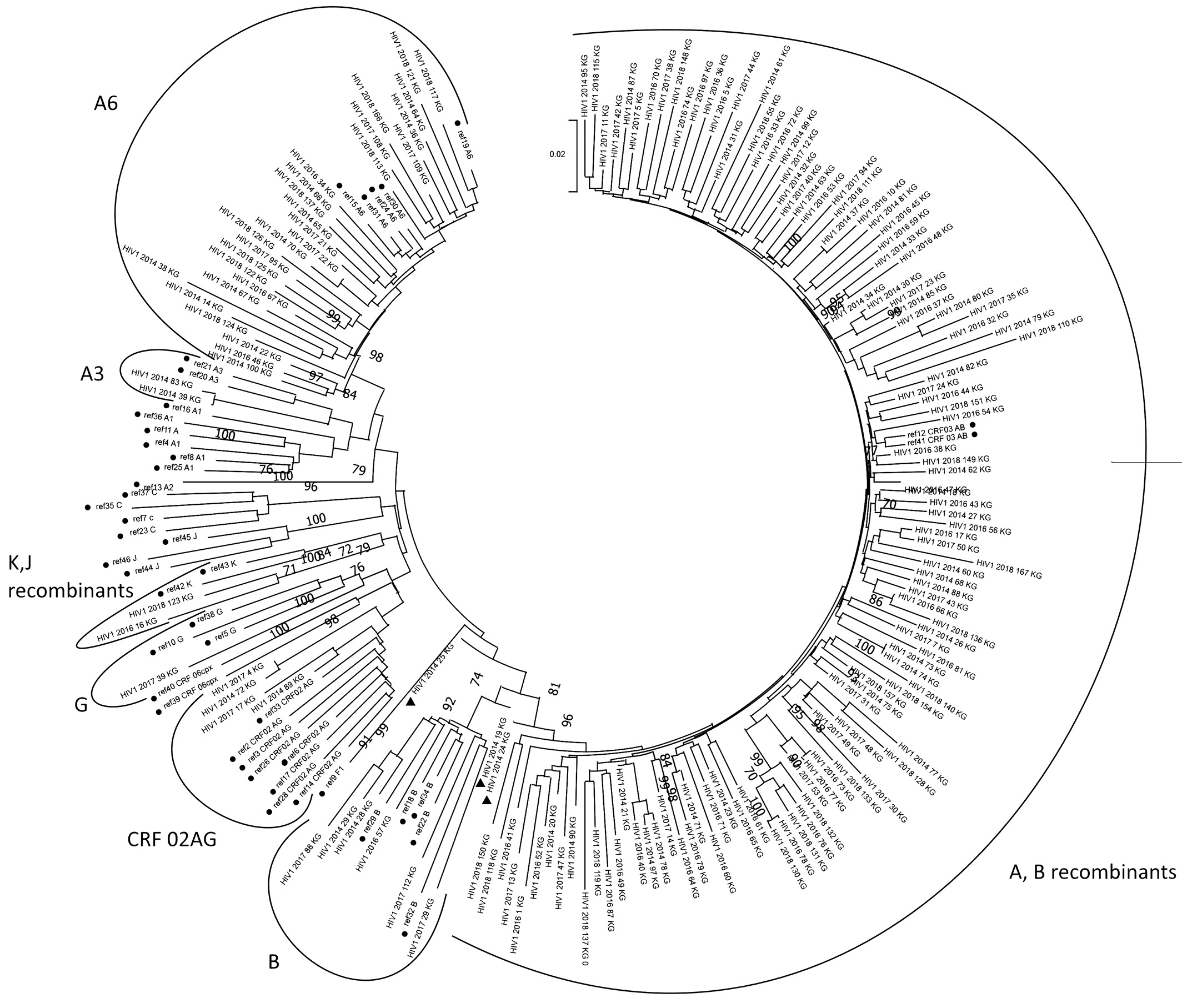
Introduction. As is currently known, the epidemic process in the Kaliningrad Region was mainly associated with the spread of the recombinant form of HIV-1 (CRF03_AB); however, regular HIV importations from other countries and continents has created favorable conditions for emergence and spread of various recombinant forms of the virus.
The most complete information on the diversity of recombinant forms in the region is also necessary to understand the structure of drug resistance (DR).
The aim of the study was to explore the HIV-1 genetic diversity in the Kaliningrad Region.
Materials and methods. We studied 162 blood plasma samples obtained from patients from the Kaliningrad Region, both with confirmed virological failure of antiretroviral therapy (ART) and with newly diagnosed HIV infection. For reverse transcription and amplification of HIV genome fragments, diagnostic «AmpliSense HIVResist-Seq».
Results and discussion. The various recombinants between subtypes A and B (74%) were predominant in study group: recombinant was between CRF03_AB and subtype A (33.95%) and CRF03_AB-like (13.58%) were the most common. Among the “pure” subtypes of the virus, subtype A6 (16.67%). The circulation of subtypes B (3.70%) and G (1.23%) was also noted.
Ninety-six patients (59.26%) were identified with at least one mutation associated with antiretroviral (ARV) drug resistance.
Conclusion. The observed diversity of subtypes and recombinant forms of the virus implies that the new recombinants are actively emerging in the studied region, both between existing recombinant forms and “pure” subtypes, as well as between “pure” subtypes.
Full Text
##article.viewOnOriginalSite##About the authors
Alexander N. Shchemelev
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Author for correspondence.
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-3139-3674
Junior Researcher, Laboratory of Immunology and Virology of HIV Infection, St. Petersburg Pasteur Research Institute of Epidemiology and Microbiology, Federal Service for Supervision of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Russian Federation, 197101, St. PetersburgAleksandr V. Semenov
Ekaterinburg Research Institute of Viral Infections of the Federal Research Institute, State Research Center for Virology and Biotechnology “Vector” of the Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-3223-8219
Russian Federation, 620030 Ekaterinburg
Yulia V. Ostankova
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-2270-8897
Russian Federation, 197101, St. Petersburg
Ekaterina V. Naidenova
FSSI Russian Research Anti-Plague Institute «Microbe» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0001-6474-3696
Russian Federation, 410005, Saratov
Elena B. Zueva
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-0579-110X
Russian Federation, 197101, St. Petersburg
Diana E. Valutite
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0002-0931-102X
Russian Federation, 197101, St. Petersburg
Mariia A. Churina
St. Petersburg GBUZ «Botkin Clinical Infectious Diseases Hospital»
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-0424-4654
Russian Federation, 191167, St. Petersburg
Pavel A. Virolainen
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0001-5918-9395
Russian Federation, 197101, St. Petersburg
Areg A. Totolian
FBSI «Saint Petersburg Pasteur Research Institute of Epidemiology and Microbiology» of the Federal Service for Surveillance of Consumer Rights Protection and Human Welfare (Rospotrebnadzor)
Email: tvildorm@gmail.com
ORCID iD: 0000-0003-4571-8799
Russian Federation, 197101, St. Petersburg
References
- Korber B., Muldoon M., Theiler J., Gao F., Gupta R., Lapedes A., et al. Timing the ancestor of the HIV-1 pandemic strains. Science. 2000; 288(5472): 1789–96. https://doi.org/10.1126/science.288.5472.1789
- Kuiken C., Foley B., Hahn B., Marx P., McCutchan F., Mellors J.W., et al. A compilation and analysis of nucleic acid and amino acid sequences. In: Human Retroviruses and AIDS. Los Alamos; 1999.
- Los Alamos National Laboratory. HIV Circulating Recombinant Forms (CRFs). Available at: https://www.hiv.lanl.gov/content/sequence/HIV/CRFs/CRFs.html
- Simon-Loriere E., Rossolillo P., Negroni M. RNA structures, genomic organization and selection of recombinant HIV. RNA Biol. 2011; 8(2): 280–6. https://doi.org/10.4161/rna.8.2.15193
- McCutchan F.E., Carr J.K., Bajani M., Sanders-Buell E., Harry T.O., Stoeckli T.C., et al. Subtype G and multiple forms of A/G intersubtype recombinant human immunodeficiency virus type 1 in Nigeria. Virology. 1999; 254(2): 226–34. https://doi.org/10.1006/viro.1998.9505
- Montavon C., Toure-Kane C., Liegeois F., Mpoudi E., Bourgeois A., Vergne L., et al. Most env and gag subtype A HIV-1 viruses circulating in West and West Central Africa are similar to the prototype AG recombinant virus IBNG. J. Acquir. Immune. Defic. Syndr. 2000; 23(5): 363–74. https://doi.org/10.1097/00126334-200004150-00001
- Menu E., Truong T.X., Lafon M.E., Nguyen T.H., Müller-Trutwin M.C., Nguyen T.T., et al. HIV type 1 Thai subtype E is predominant in South Vietnam. AIDS Res. Hum. Retroviruses. 1996; 12(7): 629–33. https://doi.org/10.1089/aid.1996.12.629
- Piyasirisilp S., McCutchan F.E., Carr J.K., Sanders-Buell E., Liu W., Chen J., et al. A recent outbreak of human immunodeficiency virus type 1 infection in southern China was initiated by two highly homogeneous, geographically separated strains, circulating recombinant form AE and a novel BC recombinant. J. Virol. 2000; 74(23): 11286–95. https://doi.org/10.1128/jvi.74.23.11286-11295.2000
- Galetto R., Moumen A., Giacomoni V., Veron M., Charneau P., Negroni M. The structure of HIV-1 genomic RNA in the gp120 gene determines a recombination hot spot in vivo. J. Biol. Chem. 2004; 279(35): 36625–32. https://doi.org/10.1074/jbc.m405476200
- Zhuang J., Jetzt A.E., Sun G., Yu H., Klarmann G., Ron Y., et al. Human immunodeficiency virus type 1 recom-bination: rate, fidelity and putative hot spots. J. Virol. 2002; 76(22): 11273–82. https://doi.org/10.1128/jvi.76.22.11273-11282.2002
- Jetzt A.E., Yu H., Klarmann G.J., Ron Y., Preston B.D., Dougherty J.P. High rate of recombination throughout the human immunodeficiency virus type 1 genome. J. Virol. 2000; 74(3): 1234–40. https://doi.org/10.1128/jvi.74.3.1234-1240.2000
- Piantadosi A., Chohan B., Chohan V., McClelland R.S., Overbaugh J. Chronic HIV-1 infection frequently fails to protect against superinfection. PLoS Pathog 2007; 3(11): 177. https://doi.org/10.1371/journal.ppat.0030177
- Gratton S., Cheynier R., Dumaurier M.J., Oksenhendler E., Wain-Hobson S. Highly restricted spread of HIV-1 and multiply infected cells within splenic germinal centers. Proc. Natl. Acad. Sci. USA. 2000; 97(26): 14566–71. https://doi.org/10.1073/pnas.97.26.14566
- Jung A., Maier R., Vartanian J.P., Bocharov G., Jung V., Fischer U., et al. Recombination: Multiply infected spleen cells in HIV patients. Nature. 2002; 418(6894): 144. https://doi.org/10.1038/418144a
- Chen J., Dang Q., Unutmaz D., Pathak V.K., Maldarelli F., Powell D., et al. Mechanisms of nonrandom human immunodeficiency virus type 1 infection and double infection: preference in virus entry is important but is not the sole factor. J. Virol 2005; 79(7): 4140–9. https://doi.org/10.1128/jvi.79.7.4140-4149.2005
- Bobkov A.F., Kazennova E.V., Selimova L.M., Khanina T.A., Ryabov G.S., Bobkova M.R., et al. Temporal trends in the HIV-1 epidemic in Russia: predominance of subtype A. J. Med. Virol. 2004; 74(2): 191–6. https://doi.org/10.1002/jmv.20177
- Schlösser M., Kartashev V.V., Mikkola V.H., Shemshura A., Saukhat S., Kolpakov D., et al. HIV-1 sub-subtype A6: Settings for normalised identification and molecular epidemiology in the Southern Federal District, Russia. Viruses. 2020; 12(4): 475. https://doi.org/10.3390/v12040475
- Liitsola K., Tashkinova I., Laukkanen T., Korovina G., Smolskaja T., Momot O., et al. HIV-1 genetic subtype A/B recombinant strain causing an explosive epidemic in injecting drug users in Kaliningrad. AIDS. 1998; 12(14): 1907–19. https://doi.org/10.1097/00002030-199814000-00023
- Lebedev A., Pasechnik O., Ozhmegova E., Antonova A., Blokh A., Grezina L., et al. Prevalence and spatiotemporal dynamics of HIV-1 Circulating Recombinant Form 03_AB (CRF03_AB) in the Former Soviet Union countries. PLoS One. 2020; 15(10): e0241269. https://doi.org/10.1371/journal.pone.0241269
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 2016; 33(7): 1870–4. https://doi.org/10.1093/molbev/msw054
- Stanford University. HIV Drug Resistance Database. REGA HIV-1 Subtyping Tool – Version 3.0. Available at: http://dbpartners.stanford.edu:8080/RegaSubtyping/stanford-hiv/typingtool/
- Stanford University. HIV Drug Resistance Database. HIVdb Program: Mutations Analysis. Available at: https://hivdb.stanford.edu/hivdb/by-patterns/
- Gottfried B. A comparative study on linear and region based diagrams. J. Spat. Inf. Sci. 2015; (10): 3–20.
- Lapovok I.A., Saleeva D.V., Kirichenko A.A., Murzakova A.V., Lopatukhin A.E., Kireev D.E. The study of dual HIV infection prevalence in Russia. Infektsionnye bolezni. 2020; 18(4): 138–48. https://doi.org/10.20953/1729-9225-2020-4-138-148 (in Russian)
- Los Alamos National Laboratory. RIP: Recombinant Identification Program. Available at: https://www.hiv.lanl.gov/content/sequence/RIP/RIP.html
- Pasechnik O.A., Blokh A.I. The prevalence of HIV recombinant forms in Russia and countries of the CIS: systematic review and metaanalysis. Infektsiya i immunitet. 2018; 8(2): 127–38. https://doi.org/10.15789/2220-7619-2018-2-127-138 (in Russian)
- Federal AIDS Center. Russian database. HIVDR in naive patients; 2020. Available at: http://www.hivrussia.info/wp-content/uploads/2020/12/2020-Rossijskaya-baza-dannyh-LU-VICH-u-naivnyh-patsientov.pdf (in Russian)
- Ostankova Yu.V., Shchemelev A.N., Zueva E.B., Churina M.A., Valutite D.E., Semenov A.V. HIV molecular epidemiology and pharmaco-resistance in patients with antiretroviral therapy failure in Arkhangelsk district. VICh infektsiya i immunosupressii. 2019; 11(4): 65–72. https://doi.org/10.22328/2077-9828-2019-11-4-79-90 (in Russian)
- Shchemelev A.N., Semenov A.V., Ostankova Yu.V., Zueva E.B., Valutite D.E., Semenova D.A., et al. Genetic diversity and drug resistance mutations of HIV-1 in Leningrad region. Zhurnal mikrobiologii, epidemiologii i immunobiologii. 2022; 99(1): 28–37. https://doi.org/10.36233/0372-9311-216 (in Russian)
- Hung M., Tokarsky E.J., Lagpacan L., Zhang L., Suo Z., Lansdon E.B. Elucidating molecular interactions of L-nucleotides with HIV-1 reverse transcriptase and mechanism of M184V-caused drug resistance. Commun. Biol. 2019; 2: 469. https://doi.org/10.1038/s42003-019-0706-x
- De Luca A., Giambenedetto S.D., Trotta M.P., Colafigli M., Prosperi M., Ruiz L., et al. Improved interpretation of genotypic changes in the HIV-1 reverse transcriptase coding region that determine the virological response to didanosine. J. Infect. Dis. 2007; 196(11): 1645–53. https://doi.org/10.1086/522231
- Lanier E.R., Givens N., Stone C., Griffin P., Gibb D., Walker S., et al. Effect of concurrent zidovudine use on the resistance pathway selected by abacavir-containing regimens. HIV Med. 2004; 5(6): 394–9. https://doi.org/10.1111/j.1468-1293.2004.00243.x
- Hu Z., Giguel F., Hatano H., Reid P., Lu J., Kuritzkes D.R. Fitness comparison of thymidine analog resistance pathways in human immunodeficiency virus type 1. J. Virol. 2006; 80(14): 7020–7. https://doi.org/10.1128/jvi.02747-05
- Ibe S., Sugiura W. Clinical significance of HIV reverse-transcriptase inhibitor-resistance mutations. Future Microbiol. 2011; 6(3): 295–315. https://doi.org/10.2217/fmb.11.7
- Xu H.T., Colby-Germinario S.P., Huang W., Oliveira M., Han Y., Quan Y., et al. Role of the K101E substitution in HIV-1 reverse transcriptase in resistance to rilpivirine and other nonnucleoside reverse transcriptase inhibitors. Antimicrob. Agents Chemother. 2013; 57(11): 5649–57. https://doi.org/10.1128/aac.01536-13
- Madruga J.V., Cahn P., Grinsztejn B., Haubrich R., Lalezari J., Mills A., et al. Efficacy and safety of TMC125 (etravirine) in treatment-experienced HIV-1-infected patients in DUET-1: 24-week results from a randomised, double-blind, placebo-controlled trial. Lancet. 2007; 370(9581): 29–38. https://doi.org/10.1016/s0140-6736(07)61047-2
- Archer R.H., Wisniewski M., Bambara R.A., Demeter L.M. The Y181C mutant of HIV-1 reverse transcriptase resistant to nonnucleoside reverse transcriptase inhibitors alters the size distribution of RNase H cleavages. Biochemistry. 2001; 40(13): 4087–95. https://doi.org/10.1021/bi002328a
- Kolomeets A.N., Varghese V., Lemey P., Bobkova M.R., Shafer R.W. A uniquely prevalent nonnucleoside reverse transcriptase inhibitor resistance mutation in Russian subtype A HIV-1 viruses. AIDS. 2014; 28(17): F1–8. https://doi.org/10.1097/qad.0000000000000485
- Ostankova Yu.V., Shchemelev A.N., Zueva E.B., Churina M.A., Valutite D.E., Semenov A.V. HIV molecular epidemiology and pharmaco-resistance in patients with antiretroviral therapy failure in Arkhangelsk district. VICh infektsiya i immunosupressii. 2019; 11(4): 65–72. https://doi.org/10.22328/2077-9828-2019-11-4-79-9 (in Russian)
- Churina M.A., Ostankova Yu.V., Semenov A.V., Nikitina N.A., Rosolovskiy A.P., Grebenkina E.V., et al. HIV-1 drug-resistance and molecular epidemiology in patients with art failure in Veliky Novgorod. VICh-infektsiya i immunosupressii. 2017; 9(1): 82–92. https://doi.org/10.22328/2077-9828-2017-9-1-82-92 (in Russian)
- Chen M., Zhu Q., Xing H., Chen H., Jin X., Dong L., et al. The characteristics of pretreatment HIV-1 drug resistance in western Yunnan, China. Epidemiol. Infect. 2020; 148: e102. https://doi.org/10.1017/s095026882000093x
- Cheung K.W., Peng Q., He L., Cai K., Jiang Q., Zhou B., et al. Rapid and simultaneous detection of major drug resistance mutations in reverse transcriptase gene for HIV-1 CRF01_AE, CRF07_BC and subtype B in China using sequenom MassARRAY® system. PLoS One. 2016; 11(4): e0153641. https://doi.org/10.1371/journal.pone.0153641
Supplementary files