Амплификационная панель NGS для секвенирования ДНК вируса гепатита В (Hepadnaviridae: Orthohepadnavirus)
- Авторы: Чанышев М.Д.1, Власенко Н.В.1, Роев Г.В.1,2, Котов И.А.2, Глущенко А.Г.1,2, Макашова В.В.1, Хафизов К.Ф.1, Акимкин В.Г.1
-
Учреждения:
- ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
- Московский физико-технический институт
- Выпуск: Том 69, № 1 (2024)
- Страницы: 65-75
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journal-vniispk.ru/0507-4088/article/view/254060
- DOI: https://doi.org/10.36233/0507-4088-212
- EDN: https://elibrary.ru/cilsjh
- ID: 254060
Цитировать
Аннотация
Введение. Гепатит В является актуальной проблемой общественного здравоохранения во всем мире. На клиническое течение заболевания, особенно на его склонность к хронизации инфекции и развитию устойчивости к терапии, значительное влияние оказывают генотип и специфические мутации вируса гепатита В (ВГВ). С учетом сохраняющейся важности эпидемиологического контроля и профилактики заболевания, существует необходимость в простом, высокочувствительном и надежном методе секвенирования полного генома ВГВ.
Цель работы. Создание и апробация амплификационной панели для полногеномного секвенирования ВГВ.
Материалы и методы. В настоящей работе мы представляем амплификационную панель NGS, предназначенную для секвенирования генома ВГВ на платформе Illumina. Панель, состоящая из 54 праймеров, разделенных на 2 пула и амплифицирующих перекрывающиеся участки генома ВГВ длиной до 300 п.н., была апробирована на 246 образцах ДНК ВГВ, выделенных из крови.
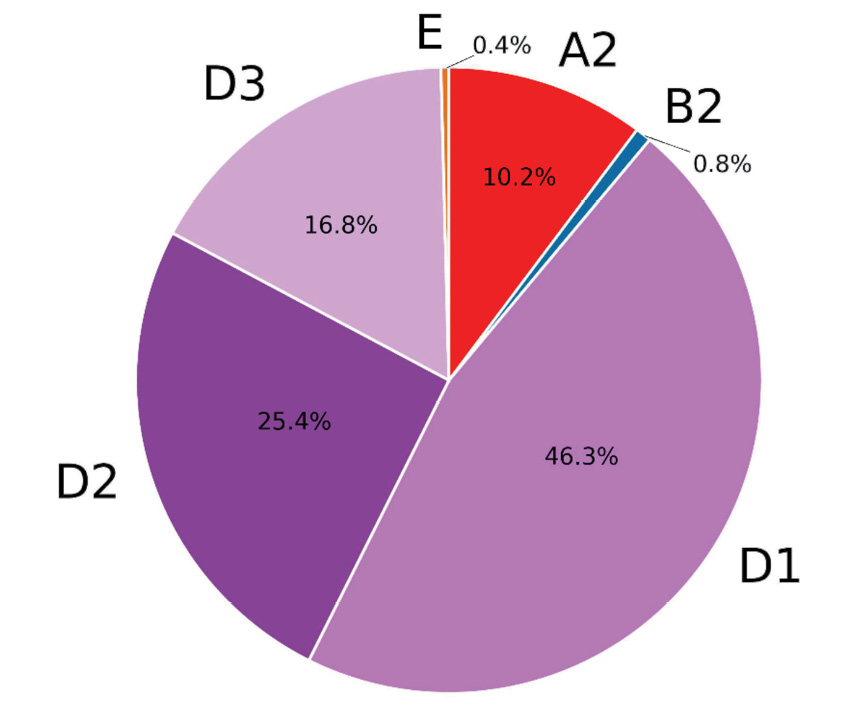
Результаты. Исследуемая выборка представляла собой широкое генотипическое разнообразие вируса, с выраженным преобладанием генотипа, характерного для Московского региона: 216 образцов были определены как генотип D, 27 – как генотип A, 2 – генотип B и 1 – генотип E. Пять образцов содержали по меньшей мере одну мутацию, связанную с устойчивостью к противовирусной терапии, в 23 образцах была найдена по меньшей мере одна мутация, связанная с ускользанием от поствакцинального ответа.
Заключение. В работе детально изложены этапы проведения полногеномного секвенирования ВГВ, приведены лабораторный протокол, нуклеотидные последовательности используемых праймеров и подход к анализу полученных данных. На примере выборки клинических образцов показана состоятельность применяемой панели. Панель для секвенирования ВГВ обладает большим потенциалом для использования в научных исследованиях, эпидемиологическом мониторинге и развитии методов персонализированной медицины.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Михаил Дамирович Чанышев
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Автор, ответственный за переписку.
Email: chanishq@gmail.com
ORCID iD: 0000-0002-6943-2915
канд. биол. наук, научный сотрудник Лаборатории геномных исследований ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. МоскваНаталья Викторовна Власенко
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: vlasenko@cmd.su
ORCID iD: 0000-0002-2388-1483
научный сотрудник Лаборатории вирусных гепатитов ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. МоскваГерман Викторович Роев
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора; Московский физико-технический институт
Email: roevherman@gmail.com
ORCID iD: 0000-0002-2353-5222
bioinformatician of the Laboratory for genomic research, Central Research Institute for Epidemiology of the Federal Service for Surveillance of Consumer Rights Protection and Human Wellbeing
Россия, 111123, г. Москва; 141701, г. ДолгопрудныйИван Андреевич Котов
Московский физико-технический институт
Email: ivan.kotov@phystech.edu
ORCID iD: 0000-0003-2416-5689
аспирант ФБМФ МФТИ
Россия, 141701, г. ДолгопрудныйАльбина Григорьевна Глущенко
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора; Московский физико-технический институт
Email: albinagluschenko@gmail.com
ORCID iD: 0009-0002-8851-8703
лаборант Лаборатории геномных исследований ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. Москва; 141701, г. ДолгопрудныйВера Васильевна Макашова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: veramakashova@yandex.ru
ORCID iD: 0000-0002-0982-3527
доктор медицинских наук, профессор, ведущий научный сотрудник клинического отдела инфекционной патологии ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. МоскваКамиль Фаридович Хафизов
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: kkhafizov@gmail.com
ORCID iD: 0000-0001-5524-0296
канд. биол. наук, заведующий Лабораторией геномных исследований ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. МоскваВасилий Геннадьевич Акимкин
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: vgakimkin@yandex.ru
ORCID iD: 0000-0003-4228-9044
академик РАН, доктор медицинских наук, профессор, директор ФБУН ЦНИИ Эпидемиологии Роспотребнадзора
Россия, 111123, г. МоскваСписок литературы
- WHO. Hepatitis B; 2022. Available at: https://www.who.int/news-room/fact-sheets/detail/hepatitis-b
- Belaiba Z., Ayouni K., Gdoura M., Kammoun Rebai W., Touzi H., Sadraoui A., et al. Whole genome analysis of hepatitis B virus before and during long-term therapy in chronic infected patients: Molecular characterization, impact on treatment and liver disease progression. Front. Microbiol. 2022; 13: 1020147. https://doi.org/10.3389/fmicb.2022.1020147
- Kramvis A. Genotypes and genetic variability of hepatitis B virus. Intervirology. 2014; 57(3-4): 141–50. https://doi.org/10.1159/000360947
- Tran T.T., Trinh T.N., Abe K. New complex recombinant genotype of hepatitis B virus identified in Vietnam. J. Virol. 2008; 82(11): 5657–63. https://doi.org/10.1128/JVI.02556-07
- Tatematsu K., Tanaka Y., Kurbanov F., Sugauchi F., Mano S., Maeshiro T., et al. A genetic variant of hepatitis B virus divergent from known human and ape genotypes isolated from a Japanese patient and provisionally assigned to new genotype J. J. Virol. 2009; 83(20): 10538–47. https://doi.org/10.1128/JVI.00462-09
- Velkov S., Ott J.J., Protzer U., Michler T. The global hepatitis B virus genotype distribution approximated from available genotyping data. Genes (Basel). 2018; 9(10): 495. https://doi.org/10.3390/genes9100495
- Sunbul M. Hepatitis B virus genotypes: global distribution and clinical importance. World J. Gastroenterol. 2014; 20(18): 5427–34. https://doi.org/10.3748/wjg.v20.i18.5427
- Delaney W.E. 4th., Yang H., Westland C.E., Das K., Arnold E., Gibbs C.S., et al. The hepatitis B virus polymerase mutation rtV173L is selected during lamivudine therapy and enhances viral replication in vitro. J. Virol. 2003; 77(21): 11833–41. https://doi.org/10.1128/jvi.77.21.11833-11841.2003
- Zhang X., Chen X., Wei M., Zhang C., Xu T., Liu L., et al. Potential resistant mutations within HBV reverse transcriptase sequences in nucleos(t)ide analogues-experienced patients with hepatitis B virus infection. Sci. Rep. 2019; 9(1): 8078. https://doi.org/10.1038/s41598-019-44604-6
- Vincenti D., Piselli P., Solmone M., D’Offizi G., Capobianchi M.R., Menzo S. Evolutionary trends of resistance mutational patterns of HBV reverse transcriptase over years (2002–2012) of different treatment regimens: The legacy of lamivudine/adefovir combination treatment. Antiviral. Res. 2017; 143: 62–8. https://doi.org/10.1016/j.antiviral.2017.03.008
- Araujo N.M., Teles S.A., Spitz N. Comprehensive analysis of clinically significant hepatitis B virus mutations in relation to genotype, subgenotype and geographic region. Front. Microbiol. 2020; 11: 616023. https://doi.org/10.3389/fmicb.2020.616023
- Liu Y., Wang C., Zhong Y., Li X., Dai J., Ren X., et al. Genotypic resistance profile of hepatitis B virus (HBV) in a large cohort of nucleos(t)ide analogue-experienced Chinese patients with chronic HBV infection. J. Viral. Hepat. 2011; 18(4): e29–39. https://doi.org/10.1111/j.1365-2893.2010.01360.x
- Carman W.F. The clinical significance of surface antigen variants of hepatitis B virus. J. Viral. Hepat. 1997; 4(Suppl. 1): 11–20. https://doi.org/10.1111/j.1365-2893.1997.tb00155.x
- Mokaya J., Vasylyeva T.I., Barnes E., Ansari M.A., Pybus O.G., Matthews P.C. Global prevalence and phylogeny of hepatitis B virus (HBV) drug and vaccine resistance mutations. J. Viral. Hepat. 2021; 28(8): 1110–20. https://doi.org/10.1111/jvh.13525
- Ababneh N.A., Sallam M., Kaddomi D., Attili A.M., Bsisu I., Khamees N., et al. Patterns of hepatitis B virus S gene escape mutants and reverse transcriptase mutations among genotype D isolates in Jordan. PeerJ. 2019; 7: e6583. https://doi.org/10.7717/peerj.6583
- Lazarevic I., Banko A., Miljanovic D., Cupic M. Immune-escape hepatitis B virus mutations associated with viral reactivation upon immunosuppression. Viruses. 2019; 11(9): 778. https://doi.org/10.3390/v11090778
- Avellón A., Echevarria J.M. Frequency of hepatitis B virus ‘a’ determinant variants in unselected Spanish chronic carriers. J. Med. Virol. 2006; 78(1): 24–36. https://doi.org/10.1002/jmv.20516
- Ma Q., Wang Y. Comprehensive analysis of the prevalence of hepatitis B virus escape mutations in the major hydrophilic region of surface antigen. J. Med. Virol. 2012; 84(2): 198–206. https://doi.org/10.1002/jmv.23183
- Araújo S.D.R., Malheiros A.P., Sarmento V.P., Nunes H.M., Freitas P.E.B. Molecular investigation of occult hepatitis B virus infection in a reference center in Northern Brazil. Braz. J. Infect. Dis. 2022; 26(3): 102367. https://doi.org/10.1016/j.bjid.2022.102367
- Zhou X., Liu D., Li Z., Zhao J., Cai S., Cao G. The mechanism of hepatitis B virus X gene in promoting hepatocellular carcinoma. J. Cancer Sci. Clin. Ther. 2022; 6(2): 222–33. https://doi.org/10.26502/jcsct.5079158
- Abdou Chekaraou M., Brichler S., Mansour W., Le Gal F., Garba A., Dény P., et al. A novel hepatitis B virus (HBV) subgenotype D (D8) strain, resulting from recombination between genotypes D and E, is circulating in Niger along with HBV/E strains. J. Gen. Virol. 2010; 91(Pt. 6): 1609–20. https://doi.org/10.1099/vir.0.018127-0
- Останкова Ю.В., Семенов А.В., Зуева Е.Б., Тотолян А.А. Первые случаи выявления вируса гепатита B субгенотипа D4 у больных хроническим, острым и скрытым вирусным гепатитом B в Российской Федерации. Молекулярная генетика, микробиология и вирусология. 2020; 38(4): 180–7. https://doi.org/10.17116/molgen202038041180 https://elibrary.ru/cuboiu
- Liu H., Shen L., Zhang S., Wang F., Zhang G., Yin Z., et al. Complete genome analysis of hepatitis B virus in Qinghai-Tibet plateau: the geographical distribution, genetic diversity, and co-existence of HBsAg and anti-HBs antibodies. Virol. J. 2020; 17(1): 75. https://doi.org/10.1186/s12985-020-01350-w
- Chen Q.Y., Jia H.H., Wang X.Y., Shi Y.L., Zhang L.J., Hu L.P., et al. Analysis of entire hepatitis B virus genomes reveals reversion of mutations to wild type in natural infection, a 15 year follow-up study. Infect. Genet. Evol. 2022; 97: 105184. https://doi.org/10.1016/j.meegid.2021.105184
- Lin S.R., Yang T.Y., Peng C.Y., Lin Y.Y., Dai C.Y., Wang H.Y., et al. Whole genome deep sequencing analysis of viral quasispecies diversity and evolution in HBeAg seroconverters. JHEP Rep. 2021; 3(3): 100254. https://doi.org/10.1016/j.jhepr.2021.100254
- HBVdb. Available at: https://hbvdb.lyon.inserm.fr/HBVdb/HBVdbIndex
- Kotov I., Saenko V., Borisova N., Kolesnikov A., Kondrasheva L., Tivanova E., et al. Effective approaches to study the genetic variability of SARS-CoV-2. Viruses. 2022; 14(9): 1855. https://doi.org/10.3390/v14091855
- Johnston A.D., Lu J., Ru K.L., Korbie D., Trau M. PrimerROC: accurate condition-independent dimer prediction using ROC analysis. Sci. Rep. 2019; 9(1): 209. https://doi.org/10.1038/s41598-018-36612-9
- Hebeler-Barbosa F., Wolf I.R., Valente G.T., Mello F.C.D.A., Lampe E., Pardini M.I.M.C., et al. A new method for next-generation sequencing of the full hepatitis B virus genome from a clinical specimen: impact for virus genotyping. Microorganisms. 2020; 8(9): 1391. https://doi.org/10.3390/microorganisms8091391
- Katoh K., Standley D.M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Mol. Biol. Evol. 2013; 30(4): 772–80. https://doi.org/10.1093/molbev/mst010
- Tamura K., Stecher G., Kumar S. MEGA11: Molecular Evolutionary Genetics Analysis version 11. Mol. Biol. Evol. 2021; 38(7): 3022–7. https://doi.org/10.1093/molbev/msab120
- Huerta-Cepas J., Serra F., Bork P. ETE 3: Reconstruction, analysis, and visualization of phylogenomic data. Mol. Biol. Evol. 2016; 33(6): 1635–8. https://doi.org/10.1093/molbev/msw046
- Al-Qahtani A.A., Al-Anazi M.R., Nazir N., Abdo A.A., Sanai F.M., Al-Hamoudi W.K., et al. The correlation between hepatitis B virus precore/core mutations and the progression of severe liver disease. Front. Cell Infect. Microbiol. 2018; 8: 355. https://doi.org/10.3389/fcimb.2018.00355
- Klushkina V.V., Kyuregyan K.K., Kozhanova T.V., Popova O.E., Dubrovina P.G., Isaeva O.V., et al. Impact of universal hepatitis В vaccination on prevalence, infection-associated morbidity and mortality, and circulation of immune escape variants in Russia. PLoS One. 2016; 11(6): e0157161. https://doi.org/10.1371/journal.pone.0157161
- Manuylov V., Chulanov V., Bezuglova L., Chub E., Karlsen A., Kyuregyan K., et al. Genetic diversity and possible origins of the hepatitis B virus in Siberian natives. Viruses. 2022; 14(11): 2465. https://doi.org/10.3390/v14112465
Дополнительные файлы