Вспомогательный белок Vpr вируса иммунодефицита человека 1-го типа (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1): особенности генетических вариантов вируса, циркулировавших на территории Московской области в 2019–2020 гг.
- Авторы: Кузнецова А.И.1, Антонова А.А.1, Макеева Е.А.2, Ким К.В.1, Мунчак Я.М.1, Меженская Е.Н.1, Орлова-Морозова Е.А.3, Пронин А.Ю.3, Прилипов А.Г.1, Галзитская О.В.1,4
-
Учреждения:
- ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
- ФГАОУ ВО «Московский политехнический университет»
- ГБУЗ МО «Центр профилактики и борьбы со СПИД»
- ФБУН «Институт теоретической и экспериментальной биофизики» Российской академии наук
- Выпуск: Том 70, № 4 (2025)
- Страницы: 324-339
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journal-vniispk.ru/0507-4088/article/view/330069
- DOI: https://doi.org/10.36233/0507-4088-296
- EDN: https://elibrary.ru/mfgcsm
- ID: 330069
Цитировать
Аннотация
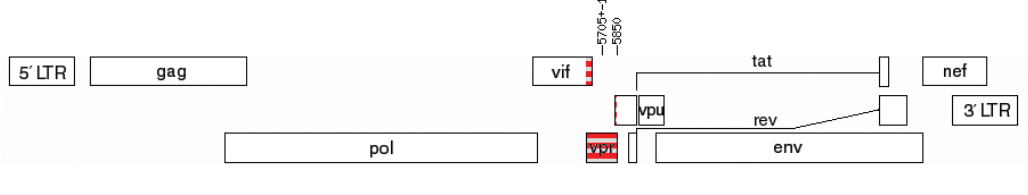
Введение. Vpr является многофункциональным вспомогательным белком ВИЧ-1, который проявляет свою активность на разных этапах жизненного цикла вируса. Олигомеризация белка является необходимым условием для проникновения Vpr в вирион и последующего его участия на ранних этапах ВИЧ-инфекции. К настоящему времени определены естественные аминокислотные замены в Vpr, ассоциированные с прогрессированием заболевания; рассматривается возможность создания средств терапии на основе Vpr.
Цель исследования – изучение особенностей Vpr у наиболее распространенных генетических вариантов ВИЧ-1, циркулировавших на территории Московской области в 2019–2020 гг.
Материалы и методы. Исследовали образцы ВИЧ-1, полученные от 231 пациента ГБУЗ МО «Центр профилактики и борьбы со СПИД» в период 2019–2020 гг. с разными стадиями заболевания, по схеме: экстракция провирусной ДНК, амплификация гена vpr, секвенирование, анализ полученных данных. Изучали консенсусные последовательности Vpr наиболее распространенных в России генетических вариантов и их пространственные структуры, вариабельность Vpr вариантов ВИЧ-1 суб-субтипа A6 (Vpr-A6) у пациентов с разными стадиями заболевания.
Результаты. Особенности Vpr различных генетических вариантов ВИЧ-1, циркулирующих в России, могут влиять на формирование его олигомерных форм. Не было выявлено ни одного сайта со статистически значимыми различиями в частоте встречаемости аминокислотных замен у пациентов с разными стадиями заболевания.
Заключение. Белки Vpr генетических вариантов ВИЧ-1, циркулирующих в России, могут иметь различия в функциональных свойствах. Варианты Vpr-A6 имели низкую вариабельность у пациентов с разными стадиями заболевания, в связи с чем Vpr-А6 может рассматриваться как объект для создания средств терапии.
Ключевые слова
Полный текст
Открыть статью на сайте журналаОб авторах
Анна Игоревна Кузнецова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: a-myznikova@list.ru
ORCID iD: 0000-0001-5299-3081
канд. биол. наук, заведующая лабораторией вирусов лейкозов, ведущий научный сотрудник Института вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваАнастасия Александровна Антонова
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Email: aantonova1792@gmail.com
ORCID iD: 0000-0002-9180-9846
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов, Институт вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваЕкатерина Александровна Макеева
ФГАОУ ВО «Московский политехнический университет»
Email: makeevakaty13@gmail.com
ORCID iD: 0009-0005-7085-3361
студентка факультета химической технологии и биотехнологии
Россия, 107023, г. МоскваКристина Вячеславовна Ким
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Email: kimsya99@gmail.com
ORCID iD: 0000-0002-4150-2280
младший научный сотрудник лаборатории вирусов лейкозов, Институт вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваЯна Михайловна Мунчак
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Email: yana_munchak@mail.ru
ORCID iD: 0000-0002-4792-8928
младший научный сотрудник лаборатории вирусов лейкозов, Институт вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваЕкатерина Никитична Меженская
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Email: belokopytova.01@mail.ru
ORCID iD: 0000-0002-3110-0843
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов, Институт вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваЕлена Александровна Орлова-Морозова
ГБУЗ МО «Центр профилактики и борьбы со СПИД»
Email: orlovamorozova@gmail.com
ORCID iD: 0000-0003-2495-6501
канд. мед. наук, заведующая амбулаторно-поликлиническим отделением
Россия, 140053, г. Котельники, Московская областьАлександр Юрьевич Пронин
ГБУЗ МО «Центр профилактики и борьбы со СПИД»
Email: alexanderp909@gmail.com
ORCID iD: 0000-0001-9268-4929
канд. мед. наук, главный врач
Россия, 140053, г. Котельники, Московская областьАлексей Геннадьевич Прилипов
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России
Email: a_prilipov@mail.ru
ORCID iD: 0000-0001-8755-1419
д-р биол. наук, ведущий научный сотрудник, заведующий лабораторией молекулярной генетики Института вирусологии им. Д.И. Ивановского
Россия, 123098, г. МоскваОксана Валериановна Галзитская
ФГБУ «Национальный исследовательский центр эпидемиологии и микробиологии имени Н.Ф. Гамалеи» Минздрава России; ФБУН «Институт теоретической и экспериментальной биофизики» Российской академии наук
Email: ogalzit@vega.protres.ru
ORCID iD: 0000-0002-3962-1520
д-р физ.-мат. наук, заведующая лабораторией биоинформатики, главный научный сотрудник ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи», Институт вирусологии им. Д.И. Ивановского
Россия, 123098, г. Москва; 142290, г. Пущино, Московская областьСписок литературы
- Kogan M., Rappaport J. HIV-1 accessory protein Vpr: Relevance in the pathogenesis of HIV and potential for therapeutic intervention. Retrovirology. 2011; 8: 25. https://doi.org/10.1186/1742-4690-8-25
- Morellet N., Bouaziz S., Petitjean P., Roques B.P. NMR structure of the HIV-1 regulatory protein VPR. J. Mol. Biol. 2003; 327(1): 215–27. https://doi.org/10.1016/s0022-2836(03)00060-3
- Sawaya B.E., Khalili K., Gordon J., Taube R., Amini S. Cooperative interaction between HIV-1 regulatory proteins Tat and Vpr modulates transcription of the viral genome. J. Biol. Chem. 2000; 275(45): 35209–14. https://doi.org/10.1074/jbc.M005197200
- Fritz J.V., Dujardin D., Godet J., Didier P., De Mey J., Darlix J.L., et al. HIV-1 Vpr oligomerization but not that of Gag directs the interaction between VPR and GAG. J. Virol. 2010; 84(3): 1585–96. https://doi.org/10.1128/JVI.01691-09
- Venkatachari N.J., Walker L.A., Tastan O., Le T., Dempsey T.M., Li Y., et al. Human immunodeficiency virus type 1 Vpr: oligomerization is an essential feature for its incorporation into virus particles. Virol. J. 2010; 7: 119. https://doi.org/10.1186/1743-422X-7-119
- Nodder S.B., Gummuluru S. Illuminating the role of VPR in HIV infection of myeloid cells. Front. Immunol. 2019; 10: 1606. https://doi.org/10.3389/fimmu.2019.01606
- Vanegas-Torres C.A., Schindler M. HIV-1 Vpr functions in primary CD4+ T Cells. Viruses. 2024; 16(3): 420. https://doi.org/10.3390/ v16030420
- Zhang F., Bieniasz P.D. HIV-1 VPR induces cell cycle arrest and enhances viral gene expression by depleting CCDC137. Elife. 2020; 9: e55806. https://doi.org/10.7554/eLife.55806
- Huang C.Y., Chiang S.F., Lin T.Y., Chiou S.H., Chow K.C. HIV-1 VPR triggers mitochondrial destruction by impairing Mfn2-mediated ER-mitochondria interaction. PLoS One. 2012; 7(3): e33657. https://doi.org/10.1371/journal.pone.0033657
- Zhao L., Wang S., Xu M., He Y., Zhang X., Xiong Y., et al. VPR counteracts the restriction of LAPTM5 to promote HIV-1 infection in macrophages. Nat. Commun. 2021; 12(1): 3691. https://doi.org/10.1038/s41467-021-24087-8
- Eldin P., Péron S., Galashevskaya A., Denis-Lagache N., Cogné M., Slupphaug G., et al. Impact of HIV-1 Vpr manipulation of the DNA repair enzyme UNG2 on B lymphocyte class switch recombination. J. Transl. Med. 2020; 18(1): 310. https://doi.org/10.1186/s12967-020-02478-7
- Casey Klockow L., Sharifi H.J., Wen X., Flagg M., Furuya A.K., Nekorchuk M., et al. The HIV-1 protein Vpr targets the endoribonuclease Dicer for proteasomal degradation to boost macrophage infection. Virology. 2013; 444(1-2): 191–202. https://doi.org/10.1016/j.virol.2013.06.010
- Li G., Makar T., Gerzanich V., Kalakonda S., Ivanova S., Pereira E.F.R., et al. HIV-1 VPR-induced proinflammatory response and apoptosis are mediated through the Sur1-Trpm4 channel in astrocytes. mBio. 2020; 11(6): e02939–20. https://doi.org/10.1128/mbio.02939-20
- Mukerjee R., Chang J.R., Del Valle L., Bagashev A., Gayed M.M., Lyde R.B., et al. Deregulation of microRNAs by HIV-1 VPR protein leads to the development of neurocognitive disorders. J. Biol. Chem. 2011; 286(40): 34976–85. https://doi.org/10.1074/jbc.M111.241547
- James T., Nonnemacher M.R., Wigdahl B., Krebs F.C. Defining the roles for VPR in HIV-1-associated neuropathogenesis. J. Neurovirol. 2016; 22(4): 403–15. https://doi.org/10.1007/s13365-016-0436-5
- Fabryova H., Strebel K. VPR and its cellular interaction partners: R we there yet? Cells. 2019; 8(11): 1310. https://doi.org/10.3390/cells8111310
- González M.E. The HIV-1 VPR protein: A multifaceted target for therapeutic intervention. Int. J. Mol. Sci. 2017; 18(1): 126. https://doi.org/10.3390/ijms18010126
- Dampier W., Antell G.C., Aiamkitsumrit B., Nonnemacher M.R., Jacobson J.M., Pirrone V., et al. Specific amino acids in HIV-1 Vpr are significantly associated with differences in patient neurocognitive status. J. Neurovirol. 2017; 23(1): 113–24. https://doi.org/10.1007/s13365-016-0462-3
- Hadi K., Walker L.A., Guha D., Murali R., Watkins S.C., Tarwater P., et al. Human immunodeficiency virus type 1 VPR polymorphisms associated with progressor and non-progressor individuals alter VPR-associated functions. J. Gen. Virol. 2014; 95(3): 700–11. https://doi.org/10.1099/vir.0.059576-0
- Colle J.H., Rose T., Rouzioux Ch., Garcia A. Two highly variable Vpr84 and Vpr85 residues within the HIV-1-Vpr C-terminal protein transduction domain control transductionnal activity and define a clade specific polymorphism. World Journal of AIDS. 2014; (4): 148–55. https://doi.org/10.4236/wja.2014.4201
- Hagiwara K., Ishii H., Murakami T., Takeshima S.N., Chutiwitoonchai N., Kodama E.N., et al. Synthesis of a VPR-binding derivative for use as a novel HIV-1 inhibitor. PLoS One. 2015; 10(12): e0145573. https://doi.org/10.1371/journal.pone.0145573
- Milani A., Baesi K., Agi E., Marouf G., Ahmadi M., Bolhassani A. HIV-1 accessory proteins: which one is potentially effective in diagnosis and vaccine development? Protein Pept. Lett. 2021; 28(6): 687–98. https://doi.org/10.2174/0929866528999201231213610
- Bbosa N., Kaleebu P., Ssemwanga D. HIV subtype diversity worldwide. Curr. Opin. HIV AIDS. 2019; 14(3): 153–60. https://doi.org/10.1097/COH.0000000000000534
- Антонова А.А., Кузнецова А.И., Ожмегова Е.Н., Лебедев А.В., Казеннова Е.В., Ким К.В. и др. Генетическое разнообразие ВИЧ-1 на современном этапе эпидемии в Российской Федерации: увеличение распространенности рекомбинантных форм. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 61–72. https://doi.org/10.22328/2077-9828-2023-15-3-61-72 https://elibrary.ru/tpwttn
- Antonova A., Kazennova E., Lebedev A., Ozhmegova E., Kuznetsova A., Tumanov A., et al. Recombinant forms of HIV-1 in the last decade of the epidemic in the Russian Federation. Viruses. 2023; 15(12): 2312. https://doi.org/10.3390/v15122312
- Maksimenko L.V., Sivay M.V., Totmenin A.V., Shvalov A.N., Skudarnov S.E., Ostapova T.S., et al. Novel HIV-1 A6/B recombinant forms (CRF133_A6B and URF_A6/B) circulating in Krasnoyarsk region, Russia. J. Infect. 2022; 85(6): 702–69. https://doi.org/10.1016/j.jinf.2022.10.001
- Halikov M.R., Ekushov V.E., Totmenin A.V., Gashnikova N.M., Antonets M.E., Tregubchak T.V., et al. Identification of a novel HIV-1 circulating recombinant form CRF157_A6C in Primorsky Territory, Russia. J. Infect. 2024; 88(2): 180–2. https://doi.org/10.1016/j.jinf.2023.11.005
- Miller S.A., Dykes D.D., Polesky H.F. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic. Acids. Res. 1988; 16(3): 1215. https://doi.org/10.1093/nar/16.3.1
- Struck D., Lawyer G., Ternes A.M., Schmit J.C., Bercoff D.P. COMET: adaptive context-based modeling for ultrafast HIV-1 subtype identification. Nucleic Acids Res. 2014; 42(18): e144. https://doi.org/10.1093/nar/gku739
- Schultz A.K., Bulla I., Abdou-Chekaraou M., Gordien E., Morgenstern B., Zoaulim F., et al. jpHMM: recombination analysis in viruses with circular genomes such as the hepatitis B virus. Nucleic Acids Res. 2012; 40(Web Server issue): W193–8. https://doi.org/10.1093/nar/gks414
- Nguyen L.T., Schmidt H.A., von Haeseler A., Minh B.Q. IQ-TREE: a fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol. Biol. Evol. 2015; 32(1): 268–74. https://doi.org/10.1093/molbev/msu300
- Larsson A. AliView: a fast and lightweight alignment viewer and editor for large datasets. Bioinformatics. 2014; 30(22): 3276–8. https://doi.org/10.1093/bioinformatics/btu531
- Darriba D., Taboada G.L., Doallo R., Posada D. jModelTest 2: more models, new heuristics and parallel computing. Nat. Methods. 2012; 9(8): 772. https://doi.org/10.1038/nmeth.2109
- Letunic I., Bork P. Interactive Tree Of Life (iTOL) v5: an online tool for phylogenetic tree display and annotation. Nucleic Acids Res. 2021; 49(W1): W293–6. https://doi.org/10.1093/nar/gkab301
- Lobanov M.Y., Sokolovskiy I.V., Galzitskaya O.V. IsUnstruct: prediction of the residue status to be ordered or disordered in the protein chain by a method based on the Ising model. J. Biomol. Struct. Dyn. 2013; 31(10): 1034–43. https://doi.org/10.1080/07391102.2012.718529
- Jumper J., Evans R., Pritzel A., Green T., Figurnov M., Ronneberger O., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 2021; 596(7873): 583–9. https://doi.org/10.1038/s41586-021-03819-2
- Березов Т.Т., Коровкин Б.Ф. Биологическая химия. М.: Медицина; 1998.
- Lobanov M.Y., Pereyaslavets L.B., Likhachev I.V., Matkarimov B.T., Galzitskaya O.V. Is there an advantageous arrangement of aromatic residues in proteins? Statistical analysis of aromatic interactions in globular proteins. Comput. Struct. Biotechnol. J. 2021; 19: 5960–8. https://doi.org/10.1016/j.csbj.2021.10.036
- Nair M., Gettins L., Fuller M., Kirtley S., Hemelaar J. Global and regional genetic diversity of HIV-1 in 2010-21: systematic review and analysis of prevalence. Lancet Microbe. 2024; 5(11): 100912. https://doi.org/10.1016/S2666-5247(24)00151-4
- Bouman J.A., Venner C.M., Walker C., Arts E.J., Regoes R.R. Per-pathogen virulence of HIV-1 subtypes A, C and D. Proc. Biol. Sci. 2023; 290(1998): 20222572. https://doi.org/10.1098/rspb.2022.2572
- Sami Saribas A., Cicalese S., Ahooyi T.M., Khalili K., Amini S., Sariyer I.K. HIV-1 Nef is released in extracellular vesicles derived from astrocytes: evidence for Nef-mediated neurotoxicity. Cell Death Dis. 2017; 8(1): e2542. https://doi.org/10.1038/cddis.2016.467
- Cafaro A., Schietroma I., Sernicola L., Belli R., Campagna M., Mancini F., et al. Role of HIV-1 tat protein interactions with host receptors in HIV infection and pathogenesis. Int. J. Mol. Sci. 2024; 25(3): 1704. https://doi.org/10.3390/ijms25031704
- Khan N., Geiger J.D. Role of Viral Protein U (VPU) in HIV-1 infection and pathogenesis. Viruses. 2021; 13(8): 1466. https://doi.org/10.3390/v13081466
- Ruiz A.P., Ajasin D.O., Ramasamy S., DesMarais V., Eugenin E.A., Prasad V.R. A naturally occurring polymorphism in the HIV-1 tat basic domain inhibits uptake by bystander cells and leads to reduced neuroinflammation. Sci. Rep. 2019; 9(1): 3308. https://doi.org/10.1038/s41598-019-39531-5
- Lebedev A., Kim K., Ozhmegova E., Antonova A., Kazennova E., Tumanov A., et al. Rev protein diversity in HIV-1 group M clades. Viruses. 2024; 16(5): 759. https://doi.org/10.3390/v16050759
- Лапавок И.А. Анализ полиморфизма неструктурных областей генома варианта ВИЧ-1, доминирующего в России: Автореф. дисс. … канд. биол. наук. М.; 2009. https://elibrary.ru/nkranl
- Антонова А.А., Лебедев А.В., Ожмегова Е.Н., Шлыкова А.В., Лаповок И.А., Кузнецова А.И. Вариабельность неструктурных белков у вариантов ВИЧ-1 суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1, sub-subtype A6), циркулирующих в разных регионах Российской Федерации. Вопросы вирусологии. 2024; 69(5): 470–80. https://doi.org/10.36233/0507-4088-262 https://elibrary.ru/wbbkuq
- Murzakova A., Kireev D., Baryshev P., Lopatukhin A., Serova E., Shemshura A., et al. Molecular epidemiology of HIV-1 subtype G in the Russian Federation. Viruses. 2019; 11(4): 348. https://doi.org/10.3390/v11040348
- Щемелев А.Н., Семенов А.В., Останкова Ю.В., Найденова Е.В., Зуева Е.Б., Валутите Д.Э. и др. Генетическое разнообразие вируса иммунодефицита человека (ВИЧ-1) в Калининградской области. Вопросы вирусологии. 2022; 67(4): 310–21. https://elibrary.ru/bkswno
- Makinson A., Masquelier B., Taieb A., Peytavin G., Waldner-Combernoux A., Collin G., et al. Presence of numerous stop codons in HIV-1 reverse transcriptase proviral DNA sequences from patients with virological response to HAART. AIDS. 2006; 20(9): 1327–9. https://doi.org/10.1097/01.aids.0000232242.51286.7b
- Alidjinou E.K., Deldalle J., Robineau O., Hallaert C., Meybeck A., Huleux T., et al. Routine drug resistance testing in proviral HIV-1 DNA: Prevalence of stop codons and hypermutation, and associated factors. J. Med. Virol. 2019; 91(9): 1684–7. https://doi.org/10.1002/jmv.25474
- Бобкова М.Р. Дефектные провирусы ВИЧ: возможное участие в патогенезе ВИЧ-инфекции. Вопросы вирусологии. 2024; 69(5): 399–414. https://doi.org/10.36233/0507-4088-261 https://elibrary.ru/pselci
- Rossenkhan R., Novitsky V., Sebunya T.K., Musonda R., Gashe B.A., Essex M. Viral diversity and diversification of major non-structural genes vif, vpr, vpu, tat exon 1 and rev exon 1 during primary HIV-1 subtype C infection. PLoS One. 2012; 7(5): e35491. https://doi.org/10.1371/journal.pone.0035491
- Shen C., Gupta P., Wu H., Chen X., Huang X., Zhou Y., et al. Molecular characterization of the HIV type 1 vpr gene in infected Chinese former blood/plasma donors at different stages of diseases. AIDS Res. Hum. Retroviruses. 2008; 24(4): 661–6. https://doi.org/10.1089/aid.2007.0270
Дополнительные файлы