Особенности белка Tat не-А6-вариантов вируса иммунодефицита человека 1-го типа (Retroviridae: Lentivirus: Lentivirus humimdef1), характерных для Российской Федерации
- Авторы: Кузнецова А.И.1, Антонова А.А.1, Лебедев А.В.1, Ожмегова Е.Н.1, Шлыкова А.В.2, Лаповок И.А.2, Галзитская О.В.1,3,4
-
Учреждения:
- Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России
- ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
- ФБУН «Институт белка» Российской академии наук
- ФБУН «Институт теоретической и экспериментальной биофизики» Российской академии наук
- Выпуск: Том 69, № 6 (2024)
- Страницы: 524-534
- Раздел: ОРИГИНАЛЬНЫЕ ИССЛЕДОВАНИЯ
- URL: https://journal-vniispk.ru/0507-4088/article/view/277914
- DOI: https://doi.org/10.36233/0507-4088-272
- EDN: https://elibrary.ru/xdvhfq
- ID: 277914
Цитировать
Аннотация
Введение. Белок Tat вируса иммунодефицита человека 1-го типа (ВИЧ-1) являет транс-активатором транскрипции вирусного генома, дополнительные функции которого включают способность запускать хронический воспалительный процесс. Естественные аминокислотные замены в белке Tat могут влиять на функциональные свойства белка и на течение ВИЧ-инфекции.
Целью работы является анализ особенностей консенсусных последовательностей белка Tat не-А6-вариантов ВИЧ-1, характерных для Российской Федерации, исследование естественных полиморфизмов белка Tat варианта CRF63_02A6 и вариантов вируса субтипа В, циркулирующих на территории России.
Материалы и методы. Материалом для работы послужили полногеномные нуклеотидные последовательности ВИЧ-1 вариантов CRF63_02A6, CRF03_A6B, а также субтипа В и CRF02_AG, циркулирующих на территории России. Референсная группа была сформирована на основе последовательностей вариантов ВИЧ-1 субтипа В, циркулирующих в разных странах мира. Анализируемые последовательности преимущественно были загруженные из международной базы данных Los Alamos.
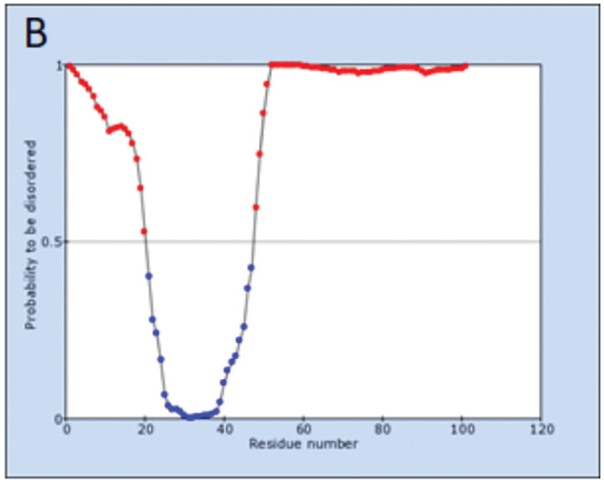
Результаты. Консенсусная последовательность CRF63_02A6 содержала наибольшее количество аминокислотных замен (31) и во вторичной структуре не содержала спирали в позициях 30‒33, однако это не привело к изменению предсказанной третичной структуры. Консенсусная последовательность CRF03_A6B содержала стоп-кодон в 87-м положении. Определены полиморфизмы вариантов ВИЧ-1 субтипа В, циркулирующих в нашей стране, и вариантов CRF63_02A6.
Заключение. Получены консенсусные последовательности белка Tat не-А6-вариантов ВИЧ-1, характерных для Российской Федерации, и определены их особенности. Замена R78G, расположенная в функционально значимом мотиве, и функционально значимая замена C31S достоверно чаще встречались у вариантов вируса субтипа В, циркулирующих на территории России, и у вариантов CRF63_02A6 соответственно, чем в референсной группе. Ограничением проведенного исследования являлась небольшая выборка последовательностей.
Полный текст
Открыть статью на сайте журналаОб авторах
Анна Игоревна Кузнецова
Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России
Автор, ответственный за переписку.
Email: a-myznikova@list.ru
ORCID iD: 0000-0001-5299-3081
канд. биол. наук, заведующая лабораторией вирусов лейкозов, ведущий научный сотрудник
Россия, 123098, г. МоскваАнастасия Александровна Антонова
Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России
Email: aantonova1792@gmail.com
ORCID iD: 0000-0002-9180-9846
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваАлексей Владимирович Лебедев
Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России
Email: lebedevalesha236@gmail.com
ORCID iD: 0000-0001-6787-9345
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваЕкатерина Никитична Ожмегова
Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России
Email: belokopytova.01@mail.ru
ORCID iD: 0000-0002-3110-0843
канд. биол. наук, научный сотрудник лаборатории вирусов лейкозов
Россия, 123098, г. МоскваАнастасия Вениаминовна Шлыкова
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: murzakova_a.v@mail.ru
ORCID iD: 0000-0002-1390-8021
научный сотрудник лаборатории диагностики и молекулярной эпидемиологии ВИЧ-инфекции
Россия, 111123, г. МоскваИлья Андреевич Лаповок
ФБУН «Центральный научно-исследовательский институт эпидемиологии» Роспотребнадзора
Email: i_lapovok@mail.ru
ORCID iD: 0000-0002-6328-1415
канд. биол. наук, старший научный сотрудник
Россия, 111123, г. МоскваОксана Валериановна Галзитская
Институт вирусологии им. Д.И. Ивановского ФГБУ «НИЦЭМ им. Н.Ф. Гамалеи» Минздрава России; ФБУН «Институт белка» Российской академии наук; ФБУН «Институт теоретической и экспериментальной биофизики» Российской академии наук
Email: ogalzit@vega.protres.ru
ORCID iD: 0000-0002-3962-1520
д-р физ.-мат. наук, заведующая лабораторией биоинформатики, главный научный сотрудник
Россия, 123098, г. Москва; 142290, Московская область, г. Пущино; 142290, Московская область, г. ПущиноСписок литературы
- German Advisory Committee Blood (Arbeitskreis Blut), Subgroup «Assessment of Pathogens Transmissible by Blood». Human Immunodeficiency Virus (HIV). Transfus. Med. Hemother. 2016; 43(3): 203–22. https://doi.org/10.1159/000445852
- Bbosa N., Kaleebu P., Ssemwanga D. HIV subtype diversity worldwide. Curr. Opin. HIV AIDS. 2019; 14(3): 153–60. https://doi.org/10.1097/COH.0000000000000534
- Кузнецова А.И. Роль полиморфизма ВИЧ-1 в патогенезе. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 26–37. https://doi.org/10.22328/2077-9828-2023-15-3-26-37 https://elibrary.ru/cwjjai
- Антонова А.А., Кузнецова А.И., Ожмегова Е.Н., Лебедев А.В., Казеннова Е.В., Ким К.В. и др. Генетическое разнообразие ВИЧ-1 на современном этапе эпидемии в Российской Федерации: увеличение распространенности рекомбинантных форм. ВИЧ-инфекция и иммуносупрессии. 2023; 15(3): 61–72. https://doi.org/10.22328/2077-9828-2023-15-3-61-72 https://elibrary.ru/tpwttn
- Кузнецова А.И., Громов К.Б., Киреев Д.Е., Шлыкова А.В., Лопатухин А.Э., Казеннова Е.В. и др. Анализ особенностей белка Tat вируса иммунодефицита человека 1 типа суб-субтипа А6 (Retroviridae: Orthoretrovirinae: Lentivirus: Human immunodeficiency virus-1). Вопросы вирусологии. 2021; 66(6): 452–63. https://doi.org/10.36233/0507-4088-83 https://elibrary.ru/cmzgyc
- Li L., Dahiya S., Kortagere S., Aiamkitsumrit B., Cunningham D., Pirrone V., et al. Impact of Tat genetic variation on HIV-1 disease. Adv. Virol. 2012; 2012: 123605. https://doi.org/10.1155/2012/123605
- Kuznetsova A., Kim K., Tumanov A., Munchak I., Antonova A., Lebedev A., et al. Features of Tat protein in HIV-1 sub-subtype A6 variants circulating in the Moscow Region, Russia. Viruses. 2023; 15(11): 2212. https://doi.org/10.3390/v15112212
- Ajasin D., Eugenin E.A. HIV-1 Tat: Role in bystander toxicity. Front. Cell. Infect. Microbiol. 2020: 10: 61. https://doi.org/10.3389/fcimb.2020.00061
- Kamori D., Ueno T. HIV-1 Tat and viral latency: what we can learn from naturally occurring sequence variations. Front. Microbiol. 2017; 8: 80. https://doi.org/10.3389/fmicb.2017.00080
- Spector C., Mele A.R., Wigdahl B., Nonnemacher M.R. Genetic variation and function of the HIV-1 Tat protein. Med. Microbiol. Immunol. 2019; 208(2): 131–69. https://doi.org/10.1007/s00430- 019-00583-z
- Ranga U., Shankarappa R., Siddappa N.B., Ramakrishna L., Nagendran R., Mahalingam M., et al. Tat protein of human immunodeficiency virus type 1 subtype C strains is a defective chemokine. J. Virol. 2004; 78(5): 2586–90. https://doi.org/10.1128/jvi.78.5.2586-2590.2004
- Ruiz A.P., Ajasin D.O., Ramasamy S., DesMarais V., Eugenin E.A., Prasad V.R. A naturally occurring polymorphism in the HIV-1 Tat basic domain inhibits uptake by bystander cells and leads to reduced neuroinflammation. Sci. Rep. 2019; 9(1): 3308. https://doi.org/10.1038/s41598-019-39531-5
- Jin H., Li D., Lin M.H., Li L., Harrich D. Tat-based therapies as an adjuvant for an HIV-1 functional cure. Viruses. 2020; 12(4): 415. https://doi.org/10.3390/v12040415
- Asamitsu K., Fujinaga K., Okamoto T. HIV Tat/P-TEFb interaction: a potential target for novel anti-HIV therapies. Molecules. 2018; 23(4): 933. https://doi.org/10.3390/molecules23040933
- Lobanov M.Y., Sokolovskiy I.V., Galzitskaya O.V. IsUnstruct: prediction of the residue status to be ordered or disordered in the protein chain by a method based on the Ising model. J. Biomol. Struct. Dyn. 2013; 31(10): 1034–43. https://doi.org/10.1080/07391102.2012.718529
- Jumper J., Evans R., Pritzel A., Green T., Figurnov M., Ronneberger O., et al. Highly accurate protein structure prediction with AlphaFold. Nature. 2021; 596(7873): 583–9. https://doi.org/10.1038/s41586-021-03819-2
- Shafer R.W., Rhee S.Y., Pillay D., Miller V., Sandstrom P., Schapiro J.M., et al. HIV-1 protease and reverse transcriptase mutations for drug resistance surveillance. AIDS. 2007; 21(2): 215–23. https://doi.org/10.1097/QAD.0b013e328011e691
- Березов Т.Т., Коровкин Б.Ф. Биологическая химия. М.: Медицина; 1998.
- Lobanov M.Y., Pereyaslavets L.B., Likhachev I.V., Matkarimov B.T., Galzitskaya O.V. Is there an advantageous arrangement of aromatic residues in proteins? Statistical analysis of aromatic interactions in globular proteins. Comput. Struct. Biotechnol. J. 2021; 19: 5960–8. https://doi.org/10.1016/j.csbj.2021.10.036
- Tee K.K., Thomson M.M., Hemelaar J. Editorial: HIV-1 genetic diversity, volume II. Front. Microbiol. 2022; 13: 1007037. https://doi.org/10.3389/fmicb.2022.1007037
- Бобкова М.Р. Генетическое разнообразие вирусов иммунодефицита человека и антиретровирусная терапия. Терапевтический архив. 2016; 88(11): 103–11. https://doi.org/10.17116/terarkh20168811103-111 https://elibrary.ru/xeaxsb
- Cilento M.E., Kirby K.A., Sarafianos S.G. Avoiding drug resistance in HIV reverse transcriptase. Chem. Rev. 2021; 121(6): 3271–96. https://doi.org/10.1021/acs.chemrev.0c00967
- Gotora P.T., Brown K., Martin D.R., van der Sluis R., Cloete R., Williams M.E. Impact of subtype C-specific amino acid variants on HIV-1 Tat-TAR interaction: insights from molecular modelling and dynamics. Virol. J. 2024; 21(1): 144. https://doi.org/10.1186/s12985-024-02419-6
- Mishra M., Vetrivel S., Siddappa N.B., Ranga U., Seth P. Clade-specific differences in neurotoxicity of human immunodeficiency virus-1 B and C Tat of human neurons: Significance of dicysteine C30C31 motif. Ann. Neurol. 2008; 63(3): 366–76. https://doi.org/10.1002/ana.21292
Дополнительные файлы