Identification of a new alphacoronavirus (Coronaviridae: Alphacoronavirus) associated with the greater horseshoe bat (Rhinolophus ferrumequinum) in the south of European part of Russia
- Authors: Lenshin S.V.1, Vishnevskaya T.V.2, Romashin A.V.3, Bulycheva Y.I.2, Vyshemirsky O.I.4, Solovyeva S.A.2, Gitelman A.K.2, Pazilin A.S.2, Lvov D.K.2, Hu B.5, Shi Z.5,6, Alkhovsky S.V.2
-
Affiliations:
- Stavropol Plague Control Research Institute of Rospotrebnadzor
- D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
- Sochi National Park
- National Research Center «Kurchatov Institute»
- Wuhan Institute of Virology, Chinese Academy of Sciences
- Guangzhou National Laboratory
- Issue: Vol 69, No 6 (2024)
- Pages: 546-557
- Section: ORIGINAL RESEARCHES
- URL: https://journal-vniispk.ru/0507-4088/article/view/277916
- DOI: https://doi.org/10.36233/0507-4088-279
- EDN: https://elibrary.ru/yiclrv
- ID: 277916
Cite item
Abstract
Introduction. Bats are recognized as primary natural reservoirs for alpha- and betacoronaviruses. The interspecies transmission of bat coronaviruses to other mammalian hosts, including livestock and humans, can lead to epidemics, epizootics, and global pandemics.
Objective. This study aims to describe coronaviruses associated with horseshoe bats (Rhinolophus spp.) in the southern regions of the European part of Russia.
Materials and methods. Fecal samples were collected from bats inhabiting caves on the southern macroslope of the Greater Caucasus (Sochi-Adler region) during 2020, 2021, and 2024. Viral genomes were detected and analyzed using high-throughput sequencing (NGS) and RT-PCR.
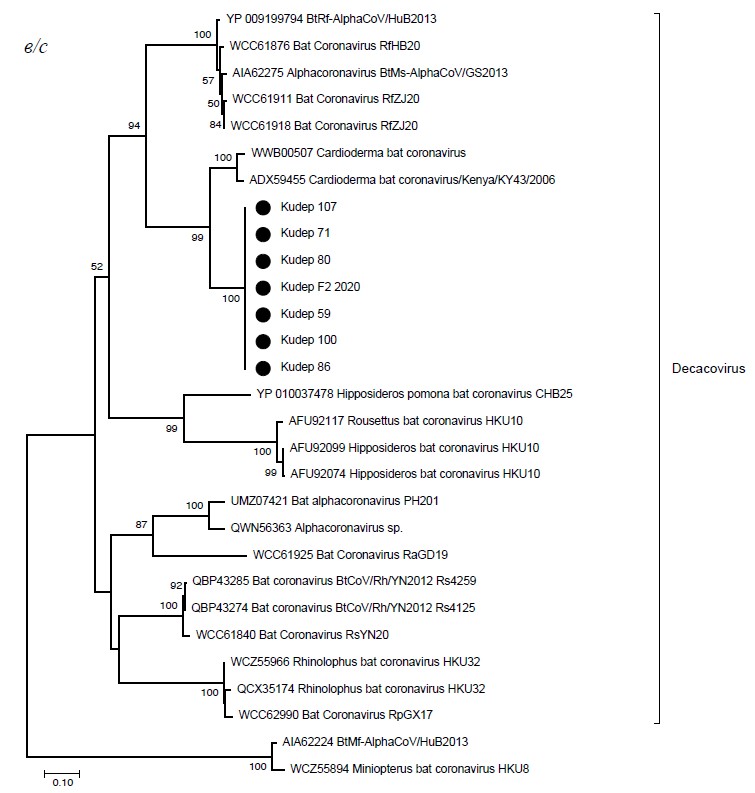
Results. A novel alphacoronavirus, designated Kudep virus (GenBank acc. # PQ649435), was identified in R. ferrumequinum. Presumably the Kudep virus represents a novel species within the subgenus Decacovirus of the genus Alphacoronavirus. The virus Showed 72% nucleotide identity to a Cardioderma bat coronavirus from Kenya and up to 67% nucleotide identity to the YN2012 virus group found in horseshoe bats in China. RT-PCR screening revealed active circulation of both Kudep virus and the previously described SARS-like betacoronavirus Khosta-1 in the study area. Infection rates in a single R. ferrumequinum colony during autumn 2021 reached 59.2% and 70.5% for Kudep and Khosta-1, respectively. Frequent co-infections with both viruses were observed in individual bats.
Conclusion. Our findings expand the understanding of the distribution of bat alphacoronaviruses and their genetic diversity. We demonstrate the presence of a persistent natural foci of two potentially zoonotic bat coronaviruses, ecologically associated with R. ferrumequinum in the southern European part of Russia.
Full Text
##article.viewOnOriginalSite##About the authors
Sergey V. Lenshin
Stavropol Plague Control Research Institute of Rospotrebnadzor
Email: lenshin-s@mail.ru
ORCID iD: 0000-0001-6815-2869
researcher at the bacteriological laboratory, Sochi department
Russian Federation, 354000, SochiTatyana V. Vishnevskaya
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: t_vish77@mail.ru
ORCID iD: 0000-0002-6963-8681
researcher at the biotechnology laboratory
Russian Federation, 123098, MoscowAlexey V. Romashin
Sochi National Park
Email: romashin@sochi.com
ORCID iD: 0000-0003-4751-1484
PhD (Biology), leading researcher
Russian Federation, 354002, SochiYulia I. Bulycheva
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: boulychevayuli@yandex.ru
ORCID iD: 0000-0002-6798-7925
researcher at the laboratory of biology and indication of arboviruses
Russian Federation, 123098, MoscowOleg I. Vyshemirsky
National Research Center «Kurchatov Institute»
Email: olegvyshem@mail.ru
ORCID iD: 0000-0002-5345-8926
PhD (Medicine), leading researcher at the laboratory of infectious virology, Kurchatov Complex of Medical Primatology
Russian Federation, 354376, Veseloe village, SochiSophya A. Solovyeva
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: sassony@ya.ru
ORCID iD: 0009-0006-7021-7143
junior researcher at the laboratory of biology and indication of arboviruses
Russian Federation, 123098, MoscowAsya K. Gitelman
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: gitelman_ak@mail.ru
ORCID iD: 0000-0002-8410-2332
leading researcher at the biotechnology laboratory
Russian Federation, 123098, MoscowAlexey S. Pazilin
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: pazilin@mail.ru
ORCID iD: 0009-0002-5665-409X
senior researcher at the biotechnology laboratory
Russian Federation, 123098, MoscowDmitry K. Lvov
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Email: dk_lvov@mail.ru
ORCID iD: 0000-0001-8176-6582
RAS Full Member, Professor, Dr. Sci. (Medicine), Chief Researcher
Russian Federation, 123098, MoscowBen Hu
Wuhan Institute of Virology, Chinese Academy of Sciences
Email: huben@wh.iov.cn
ORCID iD: 0000-0001-9194-3474
PhD, researcher at the Key Laboratory of Virology and Biosafety
China, Wuhan, 430207Zheng-Li Shi
Wuhan Institute of Virology, Chinese Academy of Sciences; Guangzhou National Laboratory
Email: zlshi@wh.iov.cn
ORCID iD: 0000-0001-8089-163X
PhD, head of the Key Laboratory of Virology and Biosafety, Wuhan Institute of Virology
China, Wuhan, 430207; Guangzhou International Bio-Island, Guangzhou 51005, GuangdongSergey V. Alkhovsky
D.I. Ivanovsky institute of virology of N.F Gamaleya national research center on epidemiology and microbiology of Ministry of health of Russian Federation
Author for correspondence.
Email: s_alkhovsky@gamaleya.org
ORCID iD: 0000-0001-6913-5841
corresponding member of RAS, Dr. Sci. (Biology), head of the laboratory of biotechnology
Russian Federation, 123098, MoscowReferences
- L’vov D.K., ed. Ecology of viruses. In: Handbook of Virology. Viruses and Viral Infections of Humans and Animals [Rukovodstvo po virusologii. Virusy i virusnye infektsii cheloveka i zhivotnykh]. Moscow: MIA; 2013. (in Russian)
- Letko M., Seifert S.N., Olival K.J., Plowright R.K., Munster V.J. Bat-borne virus diversity, spillover and emergence. Nat. Rev. Microbiol. 2020; 18(8): 461–71. https://doi.org/10.1038/s41579-020-0394-z
- Grange Z.L., Goldstein T., Johnson C.K., Anthony S., Gilardi K., Daszak P., et al. Ranking the risk of animal-to-human spillover for newly discovered viruses. Proc. Natl. Acad. Sci. USA. 2021; 118(15): e2002324118. https://doi.org/10.1073/pnas.2002324118
- Forni D., Cagliani R., Clerici M., Sironi M. Molecular Evolution of Human Coronavirus Genomes. Trends Microbiol. 2016; 25(1): 35–48. https://doi.org/10.1016/j.tim.2016.09.001
- Li W., Shi Z., Yu M., Ren W., Smith C., Epstein J.H., et al. Bats are natural reservoirs of SARS-like coronaviruses. Science. 2005; 310(5748): 676–9. https://doi.org/10.1126/science.1118391
- Lvov D.K., Alkhovsky S.V. Source of the COVID-19 pandemic: ecology and genetics of coronaviruses (Betacoronavirus: Coronaviridae) SARS-CoV, SARS-CoV-2 (Subgenus Sarbecovirus), and MERS-CoV (Subgenus Merbecovirus). Voprosy Virusologii. 2020; 65(2): 62–70. https://doi.org/10.36233/0507-4088-2020-65-2-62-70 https://elibrary.ru/hnouwn (in Russian)
- Woo P.C., Lau S.K., Lam C.S., Lau C.C., Tsang A.K., Lau J.H., et al. Discovery of seven novel Mammalian and avian coronaviruses in the genus deltacoronavirus supports bat coronaviruses as the gene source of alphacoronavirus and betacoronavirus and avian coronaviruses as the gene source of gammacoronavirus and deltacoronavi. J. Virol. 2012; 86(7): 3995–4008. https://doi.org/10.1128/jvi.06540-11
- Zhou P., Fan H., Lan T., Yang X.L., Shi W.F., Zhang W., et al. Fatal swine acute diarrhoea syndrome caused by an HKU2-related coronavirus of bat origin. Nature. 2018; 556(7700): 255–8. https://doi.org/10.1038/S41586-018-0010-9
- Lenshin S.V., Romashin A.V., Vyshemirsky O.I., Lvov D.K., Alkhovsky S.V. Bats of the subtropical climate zone of the Krasnodar territory of Russia as a possible reservoir of zoonotic viral infections. Voprosy virusologii. 2021; 66(2): 112–22. https://doi.org/10.36233/0507-4088-41 https://elibrary.ru/bimauw (in Russian)
- Wang N., Luo C., Liu H., Yang X., Hu B., Zhang W., et al. Characterization of a new member of alphacoronavirus with unique genomic features in Rhinolophus bats. Viruses. 2019; 11(4): 379. https://doi.org/10.3390/v11040379
- Wu Z., Yang L., Ren X., He G., Zhang J., Yang J., et al. Deciphering the bat virome catalog to better understand the ecological diversity of bat viruses and the bat origin of emerging infectious diseases. ISME J. 2016; 10(3): 609–20. https://doi.org/10.1038/ismej.2015.138
- Lau S.K.P., Wong A.C.P., Zhang L., Luk H.K.H., Kwok J.S.L., Ahmed S.S., et al. Novel bat alphacoronaviruses in southern China support Chinese horseshoe bats as an important reservoir for potential novel coronaviruses. Viruses. 2019; 11(5): 423. https://doi.org/10.3390/v11050423
- Alkhovsky S., Lenshin S., Romashin A., Vishnevskaya T., Vyshemirsky O., Bulycheva Y., et al. SARS-like Coronaviruses in Horseshoe Bats (Rhinolophus spp.) in Russia, 2020. Viruses. 2022; 14(1): 113. https://doi.org/10.3390/v14010113
- Yashina L.N., Zhigalin A.V., Abramov S.A., Luchnikova E.M., Smetannikova N.A., Dupal T.A., et al. Coronaviruses (Coronaviridae) of bats in the northern Caucasus and south of western Siberia. Vopr. Virusol. 2024; 69(3): 255–65. https://doi.org/10.36233//0507-4088-233
- Urushadze L., Babuadze G., Shi M., Escobar L.E., Mauldin M.R., Natradeze I., et al. A cross sectional sampling reveals novel coronaviruses in bat populations of Georgia. Viruses. 2021; 14(1): 72. https://doi.org/10.3390/v14010072
- Tao Y., Tang K., Shi M., Conrardy C., Li K.S., Lau S.K., et al. Genomic characterization of seven distinct bat coronaviruses in Kenya. Virus Res. 2012; 167(1): 67–73. https://doi.org/10.1016/j.virusres.2012.04.007
- Liu D.X., Fung T.S., Chong K.K., Shukla A., Hilgenfeld R. Accessory proteins of SARS-CoV and other coronaviruses. Antiviral. Res. 2014; 109: 97–109. https://doi.org/10.1016/j.antiviral.2014.06.013
- Lau S.K., Li K.S., Tsang A.K., Shek C.T., Wang M., Choi G.K., et al. Recent transmission of a novel alphacoronavirus, bat coronavirus HKU10, from Leschenault’s rousettes to pomona leaf-nosed bats: first evidence of interspecies transmission of coronavirus between bats of different suborders. J. Virol. 2012; 86(21): 11906–18. https://doi.org/10.1128/jvi.01305-12
- Drexler J.F., Corman V.M., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral. Res. 2014; 101: 45–56. https://doi.org/10.1016/j.antiviral.2013.10.013
- Tao Y., Tang K., Shi M., Conrardy C., Li K.S., Lau S.K., et al. Genomic characterization of seven distinct bat coronaviruses in Kenya. Virus Res. 2012; 167(1): 67–73. https://doi.org/10.1016/j.virusres.2012.04.007
- Anthony S.J., Johnson C.K., Greig D.J., Kramer S., Che X., Wells H., et al. Global patterns in coronavirus diversity. Virus Evol. 2017; 3(1): vex012. https://doi.org/10.1093/ve/vex012
- Cohen L.E., Fagre A.C., Chen B., Carlson C.J., Becker D.J. Coronavirus sampling and surveillance in bats from 1996–2019: a systematic review and meta-analysis. Nat. Microbiol. 2023; 8(6): 1176–86. https://doi.org/10.1038/s41564-023-01375-1
- Bueno L.M., Rizotto L.S., Viana A.O., Silva L.M.N., de Moraes M.V.D.S., Benassi J.C., et al. High genetic diversity of alphacoronaviruses in bat species (Mammalia: Chiroptera) from the Atlantic Forest in Brazil. Transbound. Emerg. Dis. 2022; 69(5): e2863–75. https://doi.org/10.1111/tbed.14636
- Caraballo D.A., Sabio M.S., Colombo V.C., Piccirilli M.G., Vico L., Hirmas Riade S.M., et al. The role of Molossidae and Vespertilionidae in shaping the diversity of alphacoronaviruses in the Americas. Microbiol. Spectr. 2022; 10(6): e0314322. https://doi.org/10.1128/spectrum.03143-22
- Liu D.X., Fung T.S., Chong K.K., Shukla A., Hilgenfeld R. Accessory proteins of SARS-CoV and other coronaviruses. Antiviral. Res. 2014; 109: 97–109. https://doi.org/10.1016/j.antiviral.2014.06.013
- Lau S.K., Li K.S., Tsang A.K., Shek C.T., Wang M., Choi G.K., et al. Recent transmission of a novel alphacoronavirus, bat coronavirus HKU10, from Leschenault’s rousettes to pomona leaf-nosed bats: first evidence of interspecies transmission of coronavirus between bats of different suborders. J. Virol. 2012; 86(21): 11906–18. https://doi.org/10.1128/jvi.01305-12
- Fan Y., Zhao K., Shi Z.L., Zhou P. Bat Coronaviruses in China. Viruses. 2019; 11(3): 210. https://doi.org/10.3390/v11030210
- Balboni A., Palladini A., Bogliani G., Battilani M. Detection of a virus related to betacoronaviruses in Italian greater horseshoe bats. Epidemiol. Infect. 2011; 139(2): 216–9. https://doi.org/10.1017/s0950268810001147
- Wang L.F., Shi Z., Zhang S., Field H., Daszak P., Eaton B.T. Review of bats and SARS. Emerg. Infect. Dis. 2006; 12(12): 1834–40. https://doi.org/10.3201/eid1212.060401
- Drexler J.F., Corman V.M., Drosten C. Ecology, evolution and classification of bat coronaviruses in the aftermath of SARS. Antiviral. Res. 2014; 101: 45–56. https://doi.org/10.1016/j.antiviral.2013.10.013
- Lvov D.K., Shchelkanov M.Y., Alkhovsky S.V., Deryabin P.G. Zoonotic viruses of Northern Eurasia: Taxonomy and ecology. London: Academic Press, Elsevier; 2015.
- Lvov D.K., Borisevich S.V., Alkhovsky S.V., Burtseva E.I. Relevant approaches to analysis of viral genomes for biosafety. Infektsionnye bolezni: Novosti, Mneniya, Obuchenie. 2019; 8(2): 96–101. https://doi.org/10.24411/2305-3496-2019-12012 https://elibrary.ru/xbkmpl (in Russian)
Supplementary files